

Eine Reise durch die Physiologie - Wie der Körper des Menschen funktioniert

 Chromosom: χρῶμα = Farbe, σῶμα = Körper ("Farbkörper")
Chromosom: χρῶμα = Farbe, σῶμα = Körper ("Farbkörper")
Gen: γενεά = Abstammung, γένεσις = Ursprung
Euchromatin: εὖ = gut
Exon: expressed region
Granulationsgewebe: Körniges Aussehen (granum = Korn)
Heterochromatin: ἕτερος = ungleich
Intron: intragenic region
Kinetochor: κίνησις = Bewegung, χῶρος = Ort
Mitose: μίτος = (Kett-) Faden
Nukl-: nucleus = Kern, von nux, nucis = Nuss
Phänotyp: φαίνω = ich erscheine, τύπος = Gestalt
Proliferation: proles = Sprößling, Nachwuchs; ferre = tragen
tetraploid: τετραπλόος = vierfach
Transkription: transcribere = umschreiben, überschreiben
Translation: translatio = Übertragung
Zentromer: κέντρον = Mittelpunkt, μέρος = Teil
Zygote: ζυγωτός = verknüpft, von ζυγοῦν = verbinden
Unterschiede
in Genom (Erbgut),
Umweltfaktoren, epigenetischen Modifikationen, alternative Umsetzung
von Genkopien (Splicing) und graduelle Genexpression erklären die
Variabilität der physiologischen Grundausstattung des Organismus.
Wachstumsfaktoren sind essentiell für die Anregung der Zellaktivität - Beispiele:

-- Insulinähnliche (insulin-like growth factors, IGF), die ein komplexes interzelluläres
Kommunikationssystem (IGF-Achse) bilden

-- Epidermale (epidermal growth factor, EGF), sie wirken wachstumsfördernd und mitosetriggernd, u.a. bei der Wundheilung

-- TGF's (transforming growth factors) - TGF-α und TGF-β fördern Wachstum, Differenzierung und Regeneration, regulieren den Zellzyklus und induzieren Apoptose

-- Gefäßwachstumsfaktoren (vascular endothelial growth factors,
VEGF) werden durch Sauerstoffmangel und andere Faktoren
angeregt und wirken vasodilatierend, blutdrucksenkend und
permeabilitätssteigernd

-- Nervenwachstumsfaktor (nerve growth factor, NGF) - wirkt neurotroph, lässt Nervenzellen wachsen

-- Blutplättchen-Wachstumsfaktoren (platelet derived growth factors, PDGF) wirken in Embryogenese, Zellproliferation, Migration und Wundheilung

-- Fibroblasten-Wachstumsfaktoren (fibroblast growth factors, FGF) bewirken Wachstum und Differenzierung, Angiogenese, Wundheilung und embryonale Entwicklung

-- Hepatozyten-Wachstumsfaktor (hepatocyte growth factor, HGF) wirkt morphogen und motilitätssteigernd auf epitheliale, endotheliale und hämatopoetische Zellen
Bei einer Aktivierung der Proteinsynthese wird zunächst mRNA
von DNA-Sequenzen abgelesen (Transkription) und anschließend im Zytoplasma
von der Basen- in die Aminosäurensprache übersetzt (Translation),
wobei Ribosomen eine Hauptrolle spielen.
Die Transkription
ist vielfach
geregelt (Transkriptionsfaktoren), die entstehende prä-mRNA wird von
Introns befreit (splicing), alternatives splicing erlaubt
unterschiedliche
Umsetzungen des RNA-Codes. Translation bedeutet Proteinsynthese (aus Aminosäuren) anhand der mRNA-Basensequenz (erfordert Ribosomen).
Wird eine Zellteilung (Mitose) angestoßen, wird der Vorgang an bestimmten Restriktions- bzw. Kontrollpunkten
überprüft und gegebenenfalls angehalten. Dazu dienen
Kontrollfaktoren, wie das RB-Protein oder das P53-Protein, sie
überwachen die Intaktheit des Zellzyklus. In Tumorzellen kommen sie
vermehrt vor ("Tumorsuppressoren").
|
Erbinformation  Zellzyklus, Zykline, Tumorsuppressoren, Protoonkogene
Zellzyklus, Zykline, Tumorsuppressoren, Protoonkogene  Mitose
Mitose  Genom und Epigenom
Genom und Epigenom  Zellkern
Zellkern  RNA und Ribosomen, Proteinsynthese
RNA und Ribosomen, Proteinsynthese  Wachstumsfaktoren
Wachstumsfaktoren  Wundheilung
Wundheilung

Nukleoside  Histone
Histone  Chromosom, Chromatin, Chromatid
Chromosom, Chromatin, Chromatid  Replikation
Replikation  Gen
Gen  Codon
Codon  Exon
Exon  Expression
Expression  Promotor
Promotor  Alternatives Splicing
Alternatives Splicing  Enhancer
Enhancer  Phänotyp, Genotyp, Allel
Phänotyp, Genotyp, Allel  Homo / Heterozygotie
Homo / Heterozygotie  Zygote
Zygote  Transkriptionsfaktor
Transkriptionsfaktor  Induktion / Repression
Induktion / Repression  DNA-Methylierung
DNA-Methylierung  (Proto-) Onkogene
(Proto-) Onkogene  Mitose
Mitose  Mitogen
Mitogen  Zentrosom, Zentromer, Kinetochor
Zentrosom, Zentromer, Kinetochor  Genom
Genom  Epigenom
Epigenom  Transkription / Translation
Transkription / Translation  NF-κB
NF-κB  PCR
PCR
 Rb-Protein
Rb-Protein
 Nachweis von Nukleinsäuren und Proteinen
Nachweis von Nukleinsäuren und Proteinen  Core messages
Core messages
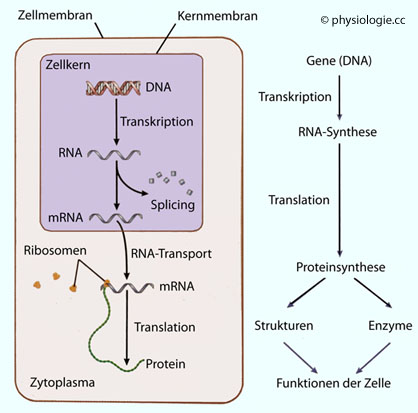
 Abbildung: Gene kontrollieren die Zellfunktionen
Abbildung: Gene kontrollieren die Zellfunktionen
Nach einer Vorlage bei Guyton and Hall, Textbook of Medical Physiology, 15th ed. Elsevier 2026
DNA
ist das Material, das die Erbinformation trägt. Die Übersetzung in RNA
nennt man Transkription ("Abschreibung"), die Umsetzung dieser
Information bei der Proteinsynthese Translation ("Übersetzung")
 Jeden Tag entstehen in unserem Körper ~10
Milliarden neue Zellen. Vermehrung der
Erbinformation, Zellteilung und Zellwachstum (Proliferation) werden kontrolliert
und gesteuert durch Enzyme wie Rezeptor-Tyrosinkinase, MAP-Kinase etc. und münden in
Transkription (Ablesung von DNA-Sequenzen, Synthese von mRNA) und
Translation (Proteinsynthese). Die entstandenen Moleküle (Enzyme,
Rezeptoren etc) vermitteln dann entsprechende Zieleffekte. Wachstumsfaktoren
spielen eine Schlüsselrolle für Erhaltung der Vitalität sowie Anregung
von Wachstum und Stoffwechsel bestimmter Zellpopulationen, Mitogene
regen die Zellteilung an.
Jeden Tag entstehen in unserem Körper ~10
Milliarden neue Zellen. Vermehrung der
Erbinformation, Zellteilung und Zellwachstum (Proliferation) werden kontrolliert
und gesteuert durch Enzyme wie Rezeptor-Tyrosinkinase, MAP-Kinase etc. und münden in
Transkription (Ablesung von DNA-Sequenzen, Synthese von mRNA) und
Translation (Proteinsynthese). Die entstandenen Moleküle (Enzyme,
Rezeptoren etc) vermitteln dann entsprechende Zieleffekte. Wachstumsfaktoren
spielen eine Schlüsselrolle für Erhaltung der Vitalität sowie Anregung
von Wachstum und Stoffwechsel bestimmter Zellpopulationen, Mitogene
regen die Zellteilung an.
Für die Synthese von Purinen und Pyrimidinen (DNA-Bausteine) benötigt der Stoffwechsel die Vitamine B9 (Folsäure) und B12 (Cobalamin). Balanciert ist die Erneuerung des Zellbestandes durch Elimination unnötiger, gealterter, veränderter oder verletzter Zellen (Apoptose).
Zellen speichern Erbinformation als linearen chemischen Code
Vorbemerkung: Die Bezeichnungen RNS und RNA bzw. DNS und DNA sind austauschbar (S = Säure, A = acid).
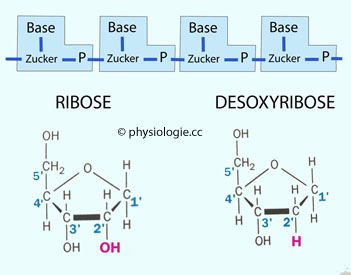
 Abbildung: Nukleinsäuren
Nach einer Vorlage in Strachan / Read: Human molecular genetics, 5th ed, CRC Press 2019
Abbildung: Nukleinsäuren
Nach einer Vorlage in Strachan / Read: Human molecular genetics, 5th ed, CRC Press 2019
Oben: Ein Nukleotid besteht aus einem Zucker (Ribose oder Desoxyribose), einem Phosphat ("P": PO43-) und einer Purin- oder Pyrimidinbase ( Abbildung unten). Aneinander gereihte Nukleotide
ergeben einen Chromatinfaden, der sich zu komplexen Strukturen
zusammenknäueln (kondensieren) kann, insbesondere in Chromosomen (s. weiter unten).
Unten: Numerierung der C-Atome
im Zuckermolekül. DNA hat (an C2) ein
Sauerstoffatom weniger im Molekül als RNA - daher die Bezeichnung
"De(s)oxy-". RNA bedeutet Ribonukleinsäure: Ribose / Desoxyribose ist
ein
Zucker, der sich jeweils über ein Phosphat (an C3 und C5: 3'-5'-Phosphodiester-Brücken) an zwei
Nachbarzuckern zu einer Kette verbindet. An C1 ist (über eine N-glykosidische Bindung) eine Base - ein Pyrimidin- oder
Purinrest - gekoppelt
Abbildung unten). Aneinander gereihte Nukleotide
ergeben einen Chromatinfaden, der sich zu komplexen Strukturen
zusammenknäueln (kondensieren) kann, insbesondere in Chromosomen (s. weiter unten).
Unten: Numerierung der C-Atome
im Zuckermolekül. DNA hat (an C2) ein
Sauerstoffatom weniger im Molekül als RNA - daher die Bezeichnung
"De(s)oxy-". RNA bedeutet Ribonukleinsäure: Ribose / Desoxyribose ist
ein
Zucker, der sich jeweils über ein Phosphat (an C3 und C5: 3'-5'-Phosphodiester-Brücken) an zwei
Nachbarzuckern zu einer Kette verbindet. An C1 ist (über eine N-glykosidische Bindung) eine Base - ein Pyrimidin- oder
Purinrest - gekoppelt

Erbinformation
wird von Zelle zu Zelle in der gesamten Biosphäre nach ähnlichen
Prinzipien übertragen. Baustein ist die DNA, und diese enthält aufeinanderfolgende Basensequenzen, von denen
viele unklare Bedeutung haben, andere als Gene wirken. Die DNA bildet
mit Begleitproteinen (Histonen und Nicht-Histonen) jeweils einen Chromatinfaden,
dieser kann in ablesbarer Form (dekondensiert: aufgelockert,
"entwirrt" - Euchromatin) vorliegen oder dicht verpackt (kondensiert,
kaum oder nicht ablesbar - Heterochromatin), insbesondere in Chromosomen (s. dort). DNA ist negativ, Histone sind positiv geladen.

 Abbildung: Struktur der DNA
Abbildung: Struktur der DNA
Nach einer Vorlage in Butler / Brown / Stephenson /
Speakman, Animal Physiology - An environmental perspective. Oxford
University Press 2021
Links: G und C sind über drei, A und T über zwei H-Brücken verknüpft.
Mitte: DNA-"Rückgrat" aus Zucker und Phosphat, Nucleobasen als
"Seitenarme". Wasserstoffbrücken stabilisieren die A/T- bzw.
G/C-Basenpaare. Die 3' (OH-Gruppe) zu 5' (Phosphat) -Richtung ist mit
Pfeilen angegeben.
Rechts: Der Radius der DNA-"Doppelwendel" beträgt 1 nm (Millionstel Millimeter)

Die DNA besteht aus zwei Ketten (Doppelhelix). Jeweils auf ein abzulesendes Gen bezogen spricht man von einem codogenen
Strang, der proteincodierend ist (die Matritze für die betreffende mRNA
abgibt) und in 5'-3'-Richtung abgelesen wird; und einem komplementären
DNA-Strang, dessen Information im Falle von DNA-Schäden zur
Rekonstruierung der kompletten DNA herangezogen werden kann.
Datenträger ist
das Chromatin, das Desoxyribonukleinsäure (DNA) und spezifische Proteine (im Verhältnis 1:4) enthält. Baustein der DNA
sind Nukleotide ; diese verbinden sich längs zu Ketten, die Basen binden komplementäre Basen (A mit T, C mit G) eines
Parallelstranges.
; diese verbinden sich längs zu Ketten, die Basen binden komplementäre Basen (A mit T, C mit G) eines
Parallelstranges.

 Abbildung: Pyrimidinbasen (oben), Purinbasen (unten)
Abbildung: Pyrimidinbasen (oben), Purinbasen (unten)
Pyrimidinbasen
leiten sich von Pyrimidin ab (aromatischer Sechserring), Purinbasen von
- aus einer 6-5-Ringkombination bestehendem - Purin

 Nukleoside bestehen aus einer Base und einer Pentose:
Nukleoside bestehen aus einer Base und einer Pentose:
Zu den Purinbasen zählen Adenin (Ade) und Guanin (Gua), zu den Pyrimidinbasen Thymin T (Thy), Cytosin (Cyt) und Uracil (Ura). Die Pentose kann Ribose (in RNA) oder Desoxyribose (in DNA) sein. Die entsprechenden Nukleoside der DNA heißen Adenosin (A), Guanosin (G), Thymidin (T) und Cytidin (C) - Purine enden auf "-sin", Pyrimidinbasen auf "-din".
Nukleotide enthalten zusätzlich Phosphat (z.B. für A: Monophosphat AMP, Diphosphat ADP, Triphosphat ATP).
Purin / Pyrimidin - Nukleosinde / Nukleotide: Abkürzungen und Vorkommen
Nach Panini SR, Medical Biochemistry, 2nd ed. 2021 (Thieme)
|
|
Base
|
Nukleosid
|
Nukleotid
(Monophosphat)
|
In:
|
Purine
|
Adenin (Ade)
|
Adenosin (A)
|
AMP
|
DNA, RNA
|
Guanin (Gua)
|
Guanosin (G)
|
GMP
|
DNA, RNA
|
Pyrimidine
|
Cytosin (Cyt)
|
Cytidin (C)
|
CMP
|
DNA, RNA
|
Thymin (Thy)
|
Thymidin (T)
|
TMP
|
DNA
|
Uracil (Ura)
|
Uridin (U)
|
UMP
|
RNA
|
Komplementarität: In der DNA-Doppelhelix bindet A jeweils an T (zwei Wasserstoffbrücken), C an G drei Wasserstoffbrücken). In der RNA ist der Bindungspartner des Adenins Uridin (statt Thymidin).
Häufigkeit: Thymin
und Adenin kommen (mit jeweils 31%, zusammen 62%) in der DNA des Menschen häufiger vor als
Cytosin und Guanin (jeweils 19%, zusammen 38%).
Eine DNA-Doppelhelix hat 2 nm Durchmesser; würde man die DNA der
Chromosomen einer diploiden menschlichen Zelle aneinanderreihen, ergäbe sich ein Faden von beinahe zwei Metern Länge
(entspricht dem 2-4.105-fachen des Zellkern-Durchmessers von ungefähr 5-10 µm).
 Über Telomere s. dort
Über Telomere s. dort

 Abbildung: Struktur eines Chromosoms
Abbildung: Struktur eines Chromosoms  Nach einer Vorlage bei Boron / Boulpaep, Medical Physiology, 1st ed., Saunders 2003
Nach einer Vorlage bei Boron / Boulpaep, Medical Physiology, 1st ed., Saunders 2003
Jedes Chromosom hat die Form eines "X" - mit einem Zentromer in der Mitte und vier als Chromatiden
bezeichneten Armen. Der Chromatinfaden liegt hier (während der Metaphase) in kondensierter
(mehrfach aufgewickelter) Form vor.
Eine DNA-Doppelhelix alleine hat eine Durchmesser von 2 nm. Die (negativ geladene) DNA ist in
Nukleosomen auf (positiv geladene) Histon-Oktamere aufgewickelt (erste Ordnung der Kondensation). Jedes Nukleosom enthält 146 DNA-Basenpaare,
eine kurze Linker-DNA (etwa 100 Basenpaare lang) verbindet Nukleosomen
untereinander. H1-Histone befestigen Linker-DNA an Nukleosomen.
N-terminale Enden der Nukleosomen-Histone (H2-H4) ragen nach außen und
sind Modifikationen (z.B. durch Methylierung, Phosphorylierung)
zugänglich (nicht dargestellt). Das beeinflusst an der betroffenen
Stelle die "Verpackung" des Chromatins und damit seine Zugänglichkeit
zur Transkription.
Im - für die Vorbereitung auf eine Mitose notwendigen - Zustand
maximaler Kondensation (wie in der Abbildung gezeigt) beträgt die Länge eines Chromosoms 0,01% der
Länge seines (theoretisch) voll "entspulten" (in die Länge gezogenen)
DNA-Fadens. Es enthält neben Histonen auch Proteine wie Condensine (für die mitotische Kondensation der Chromosomen nötig), Topoisomerasen (welche die Spiralisierung der DNA bewerkstelligen) und weitere Nicht-Histone.
Im Interphasenkern nehmen die Chromosomen definierte Orte im - in subnukleäre Zonen organisierten - Zellkern ein
 Histone sind
basische (lysin- und argininreiche) Proteine im Chromatin, welche die
Verpackung sowie die Ablesung der DNA regulieren. Auf Histon-Oktamere
ist der DNA-Strang 1,7-fach aufgewickelt und mittels Wasserstoffbrücken fixiert; diese Struktur bildet jeweils ein Nukleosom (
Histone sind
basische (lysin- und argininreiche) Proteine im Chromatin, welche die
Verpackung sowie die Ablesung der DNA regulieren. Auf Histon-Oktamere
ist der DNA-Strang 1,7-fach aufgewickelt und mittels Wasserstoffbrücken fixiert; diese Struktur bildet jeweils ein Nukleosom (
 Abbildungen),
das 147 DNA-Basenpaare enthält. Die Energie zum Aufwickeln der DNA
stammt aus elektrostatischer Anziehung zwischen negativ geladener DNA
und positiv geladenen Histonen.
Abbildungen),
das 147 DNA-Basenpaare enthält. Die Energie zum Aufwickeln der DNA
stammt aus elektrostatischer Anziehung zwischen negativ geladener DNA
und positiv geladenen Histonen.


Abbildung: Verpackung der DNA, Chromatin / Chromosom
Nach einer Vorlage in Panini SR, Medical Biochemistry, 2nd ed. 2021 (Thieme)
Links:
Die DNA-Doppelhelix wickelt sich um Paare aus 4 Histonen (H2A, H2B, H3, H4), was zur Bildung von Nukleosomen führt. H1-Histone vermitteln die Fixierung an jeweils 20 Basenpaare der DNA (die linker DNA zwischen zwei benachbarten Nukleosomen ist 50 Basenpaare lang). Nukleosomenketten (10 nm Durchmesser: 10-nm fiber) sind im Interphasenkern als Euchromatin gut abzulesen.
Mitte: Ein Chromatinfaden im Interphasenkern. In diesem Zustand (Interphase: G1, G0, S, G2) ist die DNA zugänglich, Gene sind leicht transkribierbar und die Proteinsynthese ist gut zu bewerkstelligen.
Rechts: Kondensierung des Chromatinfadens zu einem der Chromosomen im Kern der Mitosephase. Einzelne (10-nm) Nukleosomenketten sind jeweils zu dritt zu einer 30-nm fiber gruppiert (Heterochromatin).
Die 30-nm-Faser wickelt sich nochmals auf. Diese Formation ist transkriptionell inaktiv, aber stabil und
verhindert Beschädigungen der DNA im Zuge des Teilungs- und
Separierungsprozesses.
Als Kerngerüst
(Kernmatrix) bezeichnet man ein Faserwerk im Zellkern, das als
Stützstruktur für verschiedene nukleäre Funktionen dient. Legen sich
hier Chromatinfäden an, bilden sich sogenannte S/MARs (scaffold/matrix attachment regions)
- DNA-Sequenzen, an die sich die Kernmatrix heften. Zwischen solchen
Verankerungsstellen bilden sich funktionelle Einheiten, welche die
Genexpression organisieren (vgl. weiter unten)

Alleine schon das Aufwickeln der DNA auf Histonmoleküle in den
Nukleosomen ( Abbildung) reduziert die Länge des Chromatinfadens auf
ein Drittel. Dazu kommen höhere Ordnungen der Kondensation; die DNA-Fäden lassen sich
dadurch auf Mikrometerlänge "zusammenpacken". An der Organisation dieser Vorgänge sind Histone und Nicht-Histon-Proteine beteiligt, jeweils zu etwa gleicher Masse wie die entsprechende DNA.
Abbildung) reduziert die Länge des Chromatinfadens auf
ein Drittel. Dazu kommen höhere Ordnungen der Kondensation; die DNA-Fäden lassen sich
dadurch auf Mikrometerlänge "zusammenpacken". An der Organisation dieser Vorgänge sind Histone und Nicht-Histon-Proteine beteiligt, jeweils zu etwa gleicher Masse wie die entsprechende DNA.
 Chromatin ist der gesamte Komplex aus DNA, Histon- und Nicht-Histon-Proteinen, aus dem Chromosomen aufgebaut sind. Jedes Chromatid besteht aus einem DNA-Doppelstrang sowie zugehörigen Chromatin-Proteinen. Je nach Phase
des Zellzyklus kann ein Chromosom aus einem oder mehreren Chromatiden
bestehen.
Chromatin ist der gesamte Komplex aus DNA, Histon- und Nicht-Histon-Proteinen, aus dem Chromosomen aufgebaut sind. Jedes Chromatid besteht aus einem DNA-Doppelstrang sowie zugehörigen Chromatin-Proteinen. Je nach Phase
des Zellzyklus kann ein Chromosom aus einem oder mehreren Chromatiden
bestehen.
Einzelne Gene sind auch ablesbar, wenn der Großteil der DNA multipel
zusammengefaltet vorliegt, und die DNA-Histon-Aggregationen sind
dynamisch, d.h. sie ändern ständig ihre Positionen (Nukleosomengleiten).
Dieser Vorgang kann durch ATP-betriebene Proteinkomplexe - Chromatin-Umformungskomplexe - angetrieben werden; es gibt zahlreiche verschiedene, auf bestimmte Funktionen spezialisierte Umformungskomplexe. Durch die Umpositionierung der Nukleosomen werden DNA-Sequenzen neu freigelegt und damit für
Ablesung (Transkription), Reparatur- und Replikationsvorgänge zugänglich.
Die DNA wickelt sich mehrmals pro Sekunde aus einer gegebenen
Histonstruktur aus und bleibt für 10-50 Millisekunden frei zugänglich.
 Replikation (Reduplikation, DNA / RNA replication) ist
die Vervielfältigung von Erbinformation in Zellen (oder Viren). Zur
Replikation von DNA bedarf es der Mitwirkung verschiedener Proteine:
Replikation (Reduplikation, DNA / RNA replication) ist
die Vervielfältigung von Erbinformation in Zellen (oder Viren). Zur
Replikation von DNA bedarf es der Mitwirkung verschiedener Proteine:
 Topoisomerasen
starten den Vorgang des Abwickelns ("Entdröselns") der DNA durch
Aufbrechen eines Stranges und dadurch Entspannung der Helixstruktur
Topoisomerasen
starten den Vorgang des Abwickelns ("Entdröselns") der DNA durch
Aufbrechen eines Stranges und dadurch Entspannung der Helixstruktur
 Helikasen glätten die Doppelhelix nach Einwirken von Topoisomerasen an der Replikationsgabel
Helikasen glätten die Doppelhelix nach Einwirken von Topoisomerasen an der Replikationsgabel
 Einzelstrang-DNA bindende Proteine (single-strand-DNA binding proteins) erhalten die Stabilität der Replikationsgabel (Einzelstrang-DNA ist sehr empfindlich und leicht abbaubar)
Einzelstrang-DNA bindende Proteine (single-strand-DNA binding proteins) erhalten die Stabilität der Replikationsgabel (Einzelstrang-DNA ist sehr empfindlich und leicht abbaubar)
 Primasen befestigen kurze komplementäre RNA-Sequenzen (Primer) am DNA-Einzelstrang der Replikationsgabel
Primasen befestigen kurze komplementäre RNA-Sequenzen (Primer) am DNA-Einzelstrang der Replikationsgabel
 DNA-Polymerasen erzeugen entweder eine DNA-Kopie
(DNA-Polymerasen) oder RNA (RNA-Polymerasen). RNA-Polymerase I (Pol I)
transkribiert ribosomale (r) RNA, RNA-Polymerase II (Pol II) Boten (m)
RNA und die meisten kleinen nukleären (sn) RNA, RNA-Polymerase III (Pol
III) transfer (t) RNA und einige snRNA (vgl.
DNA-Polymerasen erzeugen entweder eine DNA-Kopie
(DNA-Polymerasen) oder RNA (RNA-Polymerasen). RNA-Polymerase I (Pol I)
transkribiert ribosomale (r) RNA, RNA-Polymerase II (Pol II) Boten (m)
RNA und die meisten kleinen nukleären (sn) RNA, RNA-Polymerase III (Pol
III) transfer (t) RNA und einige snRNA (vgl.  unten):
unten):
DNA-Polymerase
|
DNA → DNA
|
| RNA-Polymerase I |
DNA → rRNA
|
| RNA-Polymerase II |
DNA → mRNA, snRNA
|
| RNA-Polymerase III |
DNA → tRNA, snRNA
|
 DNA-Ligasen schweißen Knickstellen zwischen neusynthetisierter DNA zusammen.
DNA-Ligasen schweißen Knickstellen zwischen neusynthetisierter DNA zusammen.

 Gene
Gene
 sind DNA-Abschnitte, die den Bauplan einer mRNA
(messenger ribonucleic acid, Botenribonukleinsäure) und damit den eines Proteins codieren. Gene, die "abgeschrieben"
(transkribiert) werden, heißen Transkriptionseinheiten (transcription units).
sind DNA-Abschnitte, die den Bauplan einer mRNA
(messenger ribonucleic acid, Botenribonukleinsäure) und damit den eines Proteins codieren. Gene, die "abgeschrieben"
(transkribiert) werden, heißen Transkriptionseinheiten (transcription units).
Das Gen erstreckt sich von der Stelle, an der die Transkription beginnt (transcription initiation)
bis zu derjenigen, an der sie endet (transcription termination) ( Abbildung). Gene nehmen in bestimmten Chromosomen bestimmte Positionen ein - sogenannte Genloci (Einzahl: Genlocus).
Abbildung). Gene nehmen in bestimmten Chromosomen bestimmte Positionen ein - sogenannte Genloci (Einzahl: Genlocus).
Der Mensch verfügt über etwa mehr als 20.103 proteincodierende Gene (etwa gleich viel wie der 1 mm lange Fadenwurm Caenorhabditis elegans). Höhere Lebewesen nützen eine begrenzte Zahl von Genen "intelligenter"
als einfach strukturierte Vielzeller - sie sind in der Lage, gegebene
genetische Information in unterschiedlicher Art und Weise auszulesen, je nach Entwicklungsphase und
Umständen. Exakte Kontrolle der Genexpression - sowohl auf der Ebene der Transkription als auch der Translation - entscheidet über das Funktionieren unseres Genoms.
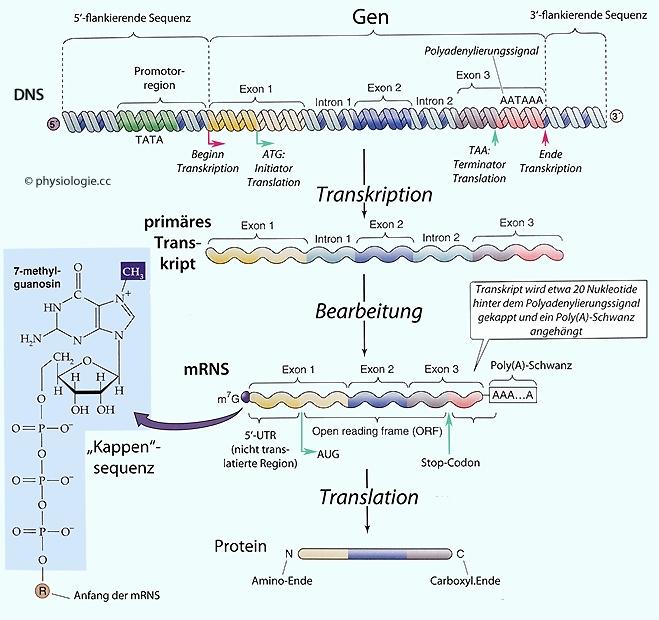
 Abbildung: Gen und Genprodukte
Nach einer Vorlage in Boron / Boulpaep: Concise Medical Physiology, Elsevier 2021
Abbildung: Gen und Genprodukte
Nach einer Vorlage in Boron / Boulpaep: Concise Medical Physiology, Elsevier 2021
Oben: Das Gen ist flankiert von DNA-Sequenzen vor (5') und hinter ihm (3' - die Bezifferung bezieht sich auf den codogenen Strang). Erste
re hat eine Promotorregion mit einer thymin- und adeninreichen Sequenz - 5'-TATAAA-3'
("TATA-Box") - knapp 30 Basenpaare vor dem Startpunkt der
Transkription (vgl. nächste  Abbildung). Das Gen-Ende ist durch ein Polyadenylierungssignal
gekennzeichnet (meist AATAAA).
Mitte: Das primäre Transkript
der RNA enthält noch Introns (die beim "Spleißen" entfernt werden) und
Exons (sie ergeben aneinandergereiht die fertige mRNA).
Unten: Die reife mRNA trägt eine "Kappe" (cap site) mit 7-Methyl-Guanosin
(m7G) und einer nichttranslatierten Sequenz (UTR, untranslated region). Dahinter befindet sich - zwischen einem Start- und einem Stop-Codon - der Leseraster (ORF, open reading frame),
der die Aminosäuresequenz des zu translatierenden Proteins codiert. Am
Ende der mRNA befindet sich ein Schwanz mit mehreren Adeninmolekülen (Poly(A) tail)
Abbildung). Das Gen-Ende ist durch ein Polyadenylierungssignal
gekennzeichnet (meist AATAAA).
Mitte: Das primäre Transkript
der RNA enthält noch Introns (die beim "Spleißen" entfernt werden) und
Exons (sie ergeben aneinandergereiht die fertige mRNA).
Unten: Die reife mRNA trägt eine "Kappe" (cap site) mit 7-Methyl-Guanosin
(m7G) und einer nichttranslatierten Sequenz (UTR, untranslated region). Dahinter befindet sich - zwischen einem Start- und einem Stop-Codon - der Leseraster (ORF, open reading frame),
der die Aminosäuresequenz des zu translatierenden Proteins codiert. Am
Ende der mRNA befindet sich ein Schwanz mit mehreren Adeninmolekülen (Poly(A) tail)
 Ein Codon ist ein kurzes Stück mRNA (ein Basentriplett), das einen Synthesebeginn (Start-Codon), eine Aminosäure oder einen Transkriptionsabbruch (Stop-Codon) codiert. tRNA-Moleküle enthalten komplimentäre Codons (Anticodons) und bringen die jeweils "passende" Aminosäure ein.
Ein Codon ist ein kurzes Stück mRNA (ein Basentriplett), das einen Synthesebeginn (Start-Codon), eine Aminosäure oder einen Transkriptionsabbruch (Stop-Codon) codiert. tRNA-Moleküle enthalten komplimentäre Codons (Anticodons) und bringen die jeweils "passende" Aminosäure ein.
Transkriptionsgeschwindigkeit an Genen nach Anregung der Zelle. Gene werden unterschiedlich rasch abgelesen: Man unterscheidet solche, die innerhalb weniger Minuten nach entsprechender Anregung der Zelle transkribiert werden (IEGs: Immediate early genes, early response genes)
- z.B. nach elektrischer Reizung oder Stimulation durch Transmitter
(u.a. bei der Ausbildung von Gedächtniselementen) bzw.
Wachstumsfaktoren - von solchen, bei denen dies Stunden dauert (delayed primary resoponse / late response genes).
Der Betrag solcher Zeitverzögerungen hängt von zahlreichen Faktoren ab,
wie Eigenschaften der Signalwege von der Zelloberfläche bis zum
Transkriptionsmechanismus, Zugang zur Promotorsequenz, Genlänge etc.
Was ist die Funktion eines Gens? Gene können funktionale RNA-Moleküle oder Proteine codieren oder die Expression funktionaler RNA-Moleküle oder von Proteinen regulieren. Die DNA des abgelesenen Gens wird zunächst 1:1 in eine entsprechende mRNA transkribiert (primäres Transkript). Dieses muss im Zellkern noch bearbeitet werden, bevor es in ein (entsprechendes) Protein übersetzt wird: Es entsteht eine "gereifte" mRNA (mature mRNA) durch Herausschneiden von RNA-Sequenzen (Introns).
 Exons
Exons  sind proteincodierende RNA-Sequenzen. Spleißen (splicing) ist das Entfernen von Introns
sind proteincodierende RNA-Sequenzen. Spleißen (splicing) ist das Entfernen von Introns  - nicht-codierenden RNA-Sequenzen - aus Prä-mRNA. (Als Exom bezeichnet man die proteincodierende Region des Genoms insgesamt - nicht zu verwechseln mit Exon.)
- nicht-codierenden RNA-Sequenzen - aus Prä-mRNA. (Als Exom bezeichnet man die proteincodierende Region des Genoms insgesamt - nicht zu verwechseln mit Exon.)
Mitochondriale DNA (mtDNA) verfügt über keine Introns. Die Transkription erfolgt mittels mitochondrialer Ribosomen unter Verwendung mitochondrialer tRNA.
 Genexpression (Exprimierung, Expression) ist
der Vorgang, der genetische Information in die Synthese funktionaler
Genprodukte umsetzt (
Genexpression (Exprimierung, Expression) ist
der Vorgang, der genetische Information in die Synthese funktionaler
Genprodukte umsetzt ( Abbildung unten). Man sagt, die Zelle exprimiert einen Stoff (z.B. Protein). Tut sie das immer, ist der Stoff ein konstitutiver Bestandteil, betreffende (konstitutive) Gene werden ständig exprimiert (und kümmern sich um grundlegende Zellfunktionen: housekeeping genes). Fakultative Gene werden hingegen nur bei Bedarf (z.B. Zellteilung) bzw. durch Anregung der Zelle - also situationsabhängig - exprimiert (inducible genes).
Abbildung unten). Man sagt, die Zelle exprimiert einen Stoff (z.B. Protein). Tut sie das immer, ist der Stoff ein konstitutiver Bestandteil, betreffende (konstitutive) Gene werden ständig exprimiert (und kümmern sich um grundlegende Zellfunktionen: housekeeping genes). Fakultative Gene werden hingegen nur bei Bedarf (z.B. Zellteilung) bzw. durch Anregung der Zelle - also situationsabhängig - exprimiert (inducible genes).
Ungleiche Genexpression bei eineiigen Zwillingen kann Unterschiede im Phänotyp bedingen.
Die Freigabe der Transkription erfolgt durch Aktivierung von Promotorsequenzen
der DNA und ist streng kontrolliert.
 Ein Promotor (promoter) ist
eine - unmittelbar vor dem 5'-Ende der betreffenden Startstelle der
Transkription gelegene - DNA-Sequenz, die notwendig ist, um ein Gen zu
exprimieren und welche die Richtung der Transkription bestimmt. Viele
Promotoren enthalten eine TATA-Box, eine thymin- und adeninreichen Sequenz, meist etwa 30 Basenpaare vor dem Ort des Transkriptionsbeginns gelegen. Promotoren sind Kombinationen aus Nukleotid-Sequenzen, meist unmittelbar vor dem betreffenden Gen (upstream) gelegen, an die RNA-Polymerase bindet und die Gen-Expression startet.
Ein Promotor (promoter) ist
eine - unmittelbar vor dem 5'-Ende der betreffenden Startstelle der
Transkription gelegene - DNA-Sequenz, die notwendig ist, um ein Gen zu
exprimieren und welche die Richtung der Transkription bestimmt. Viele
Promotoren enthalten eine TATA-Box, eine thymin- und adeninreichen Sequenz, meist etwa 30 Basenpaare vor dem Ort des Transkriptionsbeginns gelegen. Promotoren sind Kombinationen aus Nukleotid-Sequenzen, meist unmittelbar vor dem betreffenden Gen (upstream) gelegen, an die RNA-Polymerase bindet und die Gen-Expression startet.
Flankiert wird das Gen von einer Region vor (5' flanking region) und
einer nach der zu transkribierenden Sequenz (3' flanking region). Diese Regionen codieren nicht für ein Protein, sondern enthalten oft regulatorische Sequenzen (regulatory elements) - diese können die Genexpression anregen oder hemmen.
Bei Säugetieren haben mindestens 50% der Gene zwei oder mehr Promoter,
welche die Transkription über alternative Versionen eines ersten
(codierenden oder nicht codierenden) Exons (manche Gene haben ganze
Batterien davon) antreiben. Alternative Promoter können zwei Zwecken dienen: Sie können diverse - teils gewebespezifische
- regulatorische Elemente einschalten, und sie erlauben funktionell
bedeutsame Sequenzunterschiede zwischen alternativen ersten Exons (was
z.B. Protein-Isoformen mit
differenten Eigenschaften zur Folge haben kann). Die Struktur der
synthetisierten Proteine kann je nach benutztem Promoter ganz
verschieden sein - obwohl dasselbe Gen abgelesen wurde. Auf diese Weise
können die 20.103 proteincodierenden Gene des Menschen mehr als 20.104 Proteine erzeugen. Dazu kommen die Möglichkeiten des alternativen Splicings.
 Alternatives (RNA-) splicing ist eine Variante der Umsetzung eines "Vorläufer"-mRNA-Stanges (precursor messenger RNA, pre-mRNA) zu "reifer" RNA (mature messenger RNA, mRNA). Es kann
von so gut wie jedem Gen ausgehen. So können einige Exons eines Gens in die endgültige mRNA eingeschlossen sein - oder nicht -, was zur Bildung
unterschiedlicher Proteine führt.
Alternatives (RNA-) splicing ist eine Variante der Umsetzung eines "Vorläufer"-mRNA-Stanges (precursor messenger RNA, pre-mRNA) zu "reifer" RNA (mature messenger RNA, mRNA). Es kann
von so gut wie jedem Gen ausgehen. So können einige Exons eines Gens in die endgültige mRNA eingeschlossen sein - oder nicht -, was zur Bildung
unterschiedlicher Proteine führt.
Beispielsweise nehmen einige Proteine membrangebundene (mit transmembranalen
Domänen) und frei lösliche Isoformen (ohne solche Domänen) an, und
diese Isoformen können konkurrieren (und beispielsweise die Bindung von
Liganden an Rezeptoren beeinflussen).
Das Splicing kann
gewebespezifisch sein; Nervenzellen zeigen besonders intensives
alternatives Splicing (was zu verschiedenen Isoformen von Ionenkanälen und
Rezeptoren führt).
Zusammen mit Coaktivatoren beeinflussen diese die RNA-Polymeraseaktivität (vgl. oben: Die Polymerase II bildet mRNA). Um aktiv zu werden und
mRNA zu bilden, muss RNA-Polymerase II einen Transkriptionskomplex (bekannt als Präinitiationskomplex, RNA-Polymerase II Holoenzym, basal transcription machinery) mit Transkriptionsfaktoren (TF) aufbauen - Transkriptionsfaktoren A bis H ( Abbildung). TFIID enthält ein TATA-bindendes Protein.
Abbildung). TFIID enthält ein TATA-bindendes Protein.

 Abbildung: Regulatorische Sequenzen und Promotor
Abbildung: Regulatorische Sequenzen und Promotor
Nach einer Vorlage in Boron / Boulpaep: Concise Medical Physiology, Elsevier 2021
Der Transkriptionskomplex (rechts oben) konfiguriert sich am Promotor (plus der Startstelle für die Transkription),
unter dem Einfluss von Enhancern (regen die Transkription an) / Silencern (hemmen die Transkription), an die Aktivatoren /
Repressoren binden.
Pol II = RNA-Polymerase II;
kombiniert mit den Transkriptionsfaktoren A bis H (TFIIA, TFIIB etc)
bildet sie einen Transkriptionskomplex und ist so zur Anlagerung an DNA
(TFIID erkennt die TATA-Box) und zur mRNA-Synthese (Transkription) befähigt.
TATA-Box s. Text und  Abbildung oben
Abbildung oben

Die Bindung
solcher regulatorischer Faktoren verändert die Chromatinstruktur
- was sich auf die Aktivität von RNA-Polymerase und die Bindung von Transkriptionsfaktoren fördernd oder
hemmend auswirkt.
 Enhancer sind
regulatorische DNA-Elemente, die sich in einigem Abstand (bis zu 1
Megabasen upstream oder downstream) von einem Gen befinden, dessen
Expression kontrolliert werden soll. Durch - mittels eines als Cohesin bezeichneten Proteinkomplexes stabilisierte - Schleifenbildung (DNA-looping) (
Enhancer sind
regulatorische DNA-Elemente, die sich in einigem Abstand (bis zu 1
Megabasen upstream oder downstream) von einem Gen befinden, dessen
Expression kontrolliert werden soll. Durch - mittels eines als Cohesin bezeichneten Proteinkomplexes stabilisierte - Schleifenbildung (DNA-looping) ( Abbildung)
kommen sie mit ihrem Zielgen in direkten Kontakt. Enhancer bewirken den
Großteil an gewebe- bzw. zellspezifischer Genexpression, und ein
bestimmtes Gen kann zahlreiche Enhancer nutzen, die jeweils einen
bestimmten Aspekt der gewebespezifischen Genexpression steuern.
Abbildung)
kommen sie mit ihrem Zielgen in direkten Kontakt. Enhancer bewirken den
Großteil an gewebe- bzw. zellspezifischer Genexpression, und ein
bestimmtes Gen kann zahlreiche Enhancer nutzen, die jeweils einen
bestimmten Aspekt der gewebespezifischen Genexpression steuern.
Die Erbinformation (beim Menschen pro Zelle
~3.109 Basenpaare, ~2.104 proteincodierende Gene) wird
entsprechend jeweiligen
Notwendigkeiten und Erfordernissen ausgelesen. Eine typische
menschliche Zelle exprimiert davon etwa ein Drittel (~104 Gene), um
alle für ihre Tätigkeit benötigten Moleküle zur Verfügung zu haben.
Welche Gene in bestimmten Zellarten exprimiert werden (oder nicht), ist genau reguliert (gewebespezifische Genexpression). Ob bzw. wie intensiv genetische Information
realisiert wird, ist für die Zelle von entscheidender
Bedeutung: Je nach der aktuellen Situation wird sie bestimmte
Stoffwechselschritte anregen, andere eindämmen oder ganz stilllegen.

 Abbildung: Interaktion verschiedener Faktoren am Transkriptionskomplex
Nach einer Vorlage bei schoolbag.info
Abbildung: Interaktion verschiedener Faktoren am Transkriptionskomplex
Nach einer Vorlage bei schoolbag.info
Blau: DNA; rot: codierende Sequenz (Gen).
Transkription
ist die Abschreibung einer DNA-Sequenz auf Boten- Ribonukleinsäuren;
sie ist eine notwendige Vorstufe für die Proteinsynthese. Die
Transkription wird von RNA-Polymerasen vorgenommen, deren Aktivität von Zusatzfaktoren geregelt wird.
Enhancer-DNA-Sequenzen liegen upstream von der codierenden Sequenz.
An der TATA-Box (Abkürzung für die Sequenz Thymin- Adenin- Thymin- Adenin) beginnt die Assemblierung von Transkriptionsfaktoren

Die Zelle nutzt
genetische Information zur Synthese benötigter Bestandteile
(Enzyme, Fasern, Membranproteine etc) und schränkt den
Stoffwechsel auf diejenigen Teilbereiche ein, die für ihre Funktion
notwendig sind. Nur ~1,5% des gesamten Genoms sind proteincodierende Sequenzen, die aber äußerst vielfältig genutzt werden können (s. oben).
 Als Phänotyp
Als Phänotyp  bezeichnet man die (beobachtbaren) Eigenschaften und Charakteristika eines Organismus. Der Genotyp ist seine Ausstattung mit Genen (der Begriff wird meist gebraucht in Bezug auf bestimmte Allele. Ein Allel ist eine vererbte
genetische Variante einer bestimmten DNA-Sequenz innerhalb des Genoms.
Beispielsweise sind interindividuelle Unterschiede bezüglich
Blutgruppe, Augenfarbe etc. durch Allele entsprechender Gene bedingt; diese sitzen an definierten Orten bestimmter Chromosome.
bezeichnet man die (beobachtbaren) Eigenschaften und Charakteristika eines Organismus. Der Genotyp ist seine Ausstattung mit Genen (der Begriff wird meist gebraucht in Bezug auf bestimmte Allele. Ein Allel ist eine vererbte
genetische Variante einer bestimmten DNA-Sequenz innerhalb des Genoms.
Beispielsweise sind interindividuelle Unterschiede bezüglich
Blutgruppe, Augenfarbe etc. durch Allele entsprechender Gene bedingt; diese sitzen an definierten Orten bestimmter Chromosome.
 Bezüglich eines bestimmten Gens homozygot ist ein
Organismus, wenn beide Allele des betreffenden Genlocus (eines von der Mutter, eines vom Vater) eines homologen Chromosomenpaares die gleiche Nukleotidsequenz aufweisen (genetisch identisch sind). Sind die Nukleotidsequenzen der Allele
unterschiedlich, ist der Organismus bezüglich dieses Gens heterozygot
(auch wenn sich die Gene nur in einem einzigen Nukleotid
unterscheiden). Gene können in zwei oder mehr allelen Zuständen
vorliegen (20-30% der Fälle), woraus sich in der Population eine große genetische Vielfalt ergibt.
Bezüglich eines bestimmten Gens homozygot ist ein
Organismus, wenn beide Allele des betreffenden Genlocus (eines von der Mutter, eines vom Vater) eines homologen Chromosomenpaares die gleiche Nukleotidsequenz aufweisen (genetisch identisch sind). Sind die Nukleotidsequenzen der Allele
unterschiedlich, ist der Organismus bezüglich dieses Gens heterozygot
(auch wenn sich die Gene nur in einem einzigen Nukleotid
unterscheiden). Gene können in zwei oder mehr allelen Zuständen
vorliegen (20-30% der Fälle), woraus sich in der Population eine große genetische Vielfalt ergibt.
 Unter einer Zygote
Unter einer Zygote  versteht man eine (diploide) Zelle, die durch Fertilisation aus zwei (hapoliden) Gameten (einer
Eizelle und ein Spermium) entstanden ist. Bei der Fusionierung der
beiden Chromosomensätze komplettiert die Eizelle ihre Meiose 2. Die
Zygote bereitet sich anschließend auf die erste mitotische Teilung vor
und ist vorübergehend 4n-dipoid. Durch mehrfache Mitose wird das 2- und
4-Zellen-Stadium durchlaufen, anschließend entsteht eine Morula und
durch Flüssigkeitseinlagerung eine Blastula (Blastogenese).
versteht man eine (diploide) Zelle, die durch Fertilisation aus zwei (hapoliden) Gameten (einer
Eizelle und ein Spermium) entstanden ist. Bei der Fusionierung der
beiden Chromosomensätze komplettiert die Eizelle ihre Meiose 2. Die
Zygote bereitet sich anschließend auf die erste mitotische Teilung vor
und ist vorübergehend 4n-dipoid. Durch mehrfache Mitose wird das 2- und
4-Zellen-Stadium durchlaufen, anschließend entsteht eine Morula und
durch Flüssigkeitseinlagerung eine Blastula (Blastogenese).
 Transkriptionsfaktoren sind
Proteine, die sich meist an spezifische DNA-Sequenzen anlagern und die
Expression bestimmter Zielgene regulieren. Ihre Bindungsstellen nennt
man regulatorische Sequenzen bzw. Elemente. Als Cis-Element bezeichnet man einen DNA-Abschnitt, der die Ablesung eines Gens auf dem DNA-Faden verstärkt (Enhancer) oder abschwächt (Silencer). Promoter sind Cis-Elemente. An Enhancersequenzen der DNA binden als Aktivatoren bezeichnete Transkriptionsfaktoren. Andere Transkriptionsfaktoren wirken als Repressoren, sie binden an Silencersequenzen.
Transkriptionsfaktoren sind
Proteine, die sich meist an spezifische DNA-Sequenzen anlagern und die
Expression bestimmter Zielgene regulieren. Ihre Bindungsstellen nennt
man regulatorische Sequenzen bzw. Elemente. Als Cis-Element bezeichnet man einen DNA-Abschnitt, der die Ablesung eines Gens auf dem DNA-Faden verstärkt (Enhancer) oder abschwächt (Silencer). Promoter sind Cis-Elemente. An Enhancersequenzen der DNA binden als Aktivatoren bezeichnete Transkriptionsfaktoren. Andere Transkriptionsfaktoren wirken als Repressoren, sie binden an Silencersequenzen.
Bei den Transkriptionsfaktoren kennt man
 andauernd hergestellte für die permanente Grundversorgung der Zelle mit RNA, die an den unmittelbar vor der codierenden DNA-Region liegenden Kernpromotor binden (basale Transkriptionsfaktoren),
andauernd hergestellte für die permanente Grundversorgung der Zelle mit RNA, die an den unmittelbar vor der codierenden DNA-Region liegenden Kernpromotor binden (basale Transkriptionsfaktoren),
 auf regulierende weiter proximal gelegene DNA-Steuerelemente zugreifende stromaufwärts bindende (s. Abbildung),
auf regulierende weiter proximal gelegene DNA-Steuerelemente zugreifende stromaufwärts bindende (s. Abbildung),
 von außen oder durch den Zellzyklus gesteuerte induzierbare Transkriptionsfaktoren, z.B. durch Hormone - wie z,B. Steroide, Katecholamine - und deren Signalstoffe (second messenger) induzierte, oder das P53-Protein - diese Faktoren werden bedarfsmäßig aktiviert, müssen also je nach Erfordernissen "eingeschaltet" werden.
von außen oder durch den Zellzyklus gesteuerte induzierbare Transkriptionsfaktoren, z.B. durch Hormone - wie z,B. Steroide, Katecholamine - und deren Signalstoffe (second messenger) induzierte, oder das P53-Protein - diese Faktoren werden bedarfsmäßig aktiviert, müssen also je nach Erfordernissen "eingeschaltet" werden.
Beispiele für Transkriptionsfaktoren sind
 AP-1 (Activator Protein 1), steuert mit Differenzierung, Teilung und Apoptose der Zelle befasste Gene
AP-1 (Activator Protein 1), steuert mit Differenzierung, Teilung und Apoptose der Zelle befasste Gene
 ATF / CREB-Familie, eine Gruppe von Transkriptionsfaktoren, bestehend aus ATF (Activating transcription factors) und CREB, verfügen über eine DNA-Bindungsdomäne vom Leuzin-Zipper-Typ, die Proteine dimerisiert
ATF / CREB-Familie, eine Gruppe von Transkriptionsfaktoren, bestehend aus ATF (Activating transcription factors) und CREB, verfügen über eine DNA-Bindungsdomäne vom Leuzin-Zipper-Typ, die Proteine dimerisiert
 Hitzeschockfaktoren, diese regulieren die Expression von Hitzeschockproteinen
Hitzeschockfaktoren, diese regulieren die Expression von Hitzeschockproteinen
 IRF, Interferon regulatory factors, die Transkription von Interferonen regulierende Proteinediese regulieren die Expression von Hitzeschockproteinen
IRF, Interferon regulatory factors, die Transkription von Interferonen regulierende Proteinediese regulieren die Expression von Hitzeschockproteinen
 NF-κB (NF-kappa B, nuclear factor kappa-light-chain-enhancer of activated B cells)
NF-κB (NF-kappa B, nuclear factor kappa-light-chain-enhancer of activated B cells)
 SP-1 (Specificity Protein 1), ein Zinkfinger-Transkriptionsfaktor, der in zahlreiche Vorgänge (Differenzierung, Wachstum, Immunantworten etc) involviert ist
SP-1 (Specificity Protein 1), ein Zinkfinger-Transkriptionsfaktor, der in zahlreiche Vorgänge (Differenzierung, Wachstum, Immunantworten etc) involviert ist
 NF-κB ist
ein in fast allen Zellen aktiver Proteinkomplex, besonders in
proliferierenden Zellen, z.B. Immunzellen (Entwicklung, Entzündung). NF-κB bindet an eine Sequenz von ~10 Basenpaaren (κB-Motiv) und verstärkt meist die Transkription. Es kann die Aktivität von T-Zellen und NK-Zellen steigern, wenn das angeborene Immunsystem gereizt wird - über erhöhte Spiegel proinflammatorischer Zytokine
NF-κB ist
ein in fast allen Zellen aktiver Proteinkomplex, besonders in
proliferierenden Zellen, z.B. Immunzellen (Entwicklung, Entzündung). NF-κB bindet an eine Sequenz von ~10 Basenpaaren (κB-Motiv) und verstärkt meist die Transkription. Es kann die Aktivität von T-Zellen und NK-Zellen steigern, wenn das angeborene Immunsystem gereizt wird - über erhöhte Spiegel proinflammatorischer Zytokine
Promotoren sind Kombinationen aus Nukleotid-Sequenzen, meist unmittelbar vor dem betreffenden Gen (upstream) gelegen, an die RNA-Polymerase bindet und die Gen-Expression startet.
Die
genetischen Nukleotidsequenzen bestimmen also nicht nur Baupläne für
Proteine, sondern auch, wann und wie diese synthetisiert werden - also
wann und wo spezifische mRNA-Transkripte entsprechend
genregulatorischen Vorgängen gebildet werden sollen. Die Genexpression
kann auf verschiedene Weise
beeinflusst bzw. gesteuert werden ( Abbildung):
Abbildung):

 Abbildung: Phasen der Genexpression
Nach einer Vorlage in Boron / Boulpaep: Concise Medical Physiology, Elsevier 2021
Abbildung: Phasen der Genexpression
Nach einer Vorlage in Boron / Boulpaep: Concise Medical Physiology, Elsevier 2021
Beinahe alle Schritte (links) unterliegen regulatorischen Einflüssen (rechts).
Die Struktur und damit Ablesbarkeit des Chromatins (1) kann durch
von Histonen oder AcetylierungMethylierung der DNA verändert werden.
Die Transktiption muss durch Bereitstellung von DNA-Polymerase
ermöglicht werden (2); dieser Schritt ist reguliert und limitierend für
die Proteinsynthese. Das Transkript nimmt schrittweise an Länge zu (3). Löst sich die Polymerase vom DNA-Strang, hört die
Transkription auf (4).
(5) Gene können unterschiedlich gelesen werden (alternatives Spleißen:
Eine definierte DNA-Sequenz bzw. prä-RNA kann zu verschiedenen "reifen"
m-RNA und damit zu unterschiedlichen Proteinen translatiert werden. Der Großteil der Gene des Menschen unterliegt alternativem
Splicing), und unterschiedlich zu entsprechenden Zellbestandteilen realisiert werden.
(6)
Das Transkript wird in das Zytoplasma transportiert; dieser Vorgang
unterliegt einer "Qualitätskontrolle". Dann erfolgt die Proteinsynthese
(7) und schließlich der Abbau der mRNA, deren Stabilität ebenfalls
reguliert werden kann
 Induktion ist
die Ermöglichung einer Genexpression (z.B. durch ein
Stoffwechselprodukt), indem ein Aktivator (Definition s. oben) ein- bzw. Repressor
(Definition s. oben) ausgeschaltet wird; Repression bedeutet, dass umgekehrt eine Genexpression verhindert wird (indem ein Repressor aktiviert bzw. ein Aktivator ausgeschaltet wird).
Induktion ist
die Ermöglichung einer Genexpression (z.B. durch ein
Stoffwechselprodukt), indem ein Aktivator (Definition s. oben) ein- bzw. Repressor
(Definition s. oben) ausgeschaltet wird; Repression bedeutet, dass umgekehrt eine Genexpression verhindert wird (indem ein Repressor aktiviert bzw. ein Aktivator ausgeschaltet wird).
Die Physiologie des Organismus ist beeinflusst
 von Erbanlagen im klassischen Sinne (Genausstattung: Genom),
von Erbanlagen im klassischen Sinne (Genausstattung: Genom),
 Umweltfaktoren
(Wasser-, Elektrolyt-, Atemgas-, Nährstoff-, Vitamin-,
Spurenelementangebot, Temperaturprofil, Strahlung, Toxine, ..) - diese
können auch epigenetisch wirksam werden, indem sie z.B. das
Methylierungsmuster bestimmter Gene beeinflussen -, sowie
Umweltfaktoren
(Wasser-, Elektrolyt-, Atemgas-, Nährstoff-, Vitamin-,
Spurenelementangebot, Temperaturprofil, Strahlung, Toxine, ..) - diese
können auch epigenetisch wirksam werden, indem sie z.B. das
Methylierungsmuster bestimmter Gene beeinflussen -, sowie
 anderen Faktoren, wie mobilen
genetischen Elementen (Retrotransposonen),
die Kopien von sich an zufälligen Stellen ins
Genom einfügen können.
anderen Faktoren, wie mobilen
genetischen Elementen (Retrotransposonen),
die Kopien von sich an zufälligen Stellen ins
Genom einfügen können.
 DNA-Methylierung ist das Anfügen von Methylgruppen (CH3)
an ein DNA-Molekül (einen Chromatinfaden) mittels
DNA-Methyltransferasen. Dabei wird nicht die betreffende DNA-Sequenz
geändert, sondern ihre Ablesbarkeit bzw. Aktivität: DNA-Sequenzen
können ablesbar oder "maskiert" sein. Methylierung
kann Gene und/oder ihre Regulatoren (Enhancer, Promoter..)
inaktivieren. DNA-Methylierung ist der wichtigste Vorgang im Rahmen epigenetischer Veränderungen.
DNA-Methylierung ist das Anfügen von Methylgruppen (CH3)
an ein DNA-Molekül (einen Chromatinfaden) mittels
DNA-Methyltransferasen. Dabei wird nicht die betreffende DNA-Sequenz
geändert, sondern ihre Ablesbarkeit bzw. Aktivität: DNA-Sequenzen
können ablesbar oder "maskiert" sein. Methylierung
kann Gene und/oder ihre Regulatoren (Enhancer, Promoter..)
inaktivieren. DNA-Methylierung ist der wichtigste Vorgang im Rahmen epigenetischer Veränderungen.
Das Methylom
beschreibt das Methylierungsmuster des jeweiligen Genoms. Das Muster
solcher Aktivierung / Inaktivierung kann auf Tochterzellen
weitergegeben
werden (Epigenetik: Veränderung durch DNA-Methylierung oder Modifikation von Histon-Proteinen). Der Organismus unterliegt Zyklen der Methylierung und Demethylierung. "Erhaltungs-Methyltransferasen" übertragen das Methylierungsmuster
im Rahmen der Zellteilung auf Tochterstränge, d.h. es kann im Zuge der Replikation der DNA weitergegeben (vererbt) werden.
Individuelle genetische Variation ("Mutationen" ist ein weniger präziser Begriff) tritt im Laufe des Lebens unvermeidlich auf:
Die DNA in unseren Zellen unterliegt ständig Veränderungen - viele
werden repariert, andere nicht. Man kann sagen, die biologische
Ausstattung des
Individuums wird sowohl durch kausale (im Sinne der gegebenen DNA-Information) als auch Zufallsfaktoren
(z.B. Strahlung, Chemikalien) bestimmt. Solche Varianten können - je
nach Zeitpunkt in der Entwicklung - einzelne Zellen oder (wenn sie sich
teilen) Zellgruppen (Klone) unterschiedlicher Größe betreffen.
Solche DNA-Varianten sind nicht durch DNA-Reparaturmechanismen (DNA-Polymerasen, mismatch repair) korrigiert
worden und können sich - je nach Art der Variation und den jeweiligen
Umständen - als für den Organismus nachteilig, vorteilhaft (selten),
oder
belanglos erweisen. Die meisten dieser Mutationen sind unvermeidbar und
erfolgen endogen, d.h. durch spontane Fehler bei physiologischen
Reparaturvorgängen (DNA-Replikation, DNA-Reparatur, Trennung /
Rekombination von Chromosomen).
Während des gesamten Lebens kommt es zu Veränderungen der individuellen DNA (post-zygotische genetische Variation)
- wir tragen Zellen mit unterschiedlichen Genomen in uns. Das betrifft
sowohl somatische als auch Keimzellen und gilt insbesondere für B- und
T-Lymphozyten (in denen definierte Umgruppierungen der DNA-Muster stattfinden und sich eine enorme Vielfalt der Merkmale ergibt).
Wie wird der Zellzyklus kontrolliert?
Wenn sich Keimzellen vervielfältigen, tun sie das im Rahmen der Meiose:
Einem Vorgang der doppelten Zellteilung, deren Resultat die Entstehung
von vier unterschiedlichen
Tochterzellen ist, die einen jeweils einfachen Satz der 23 Chromosomen
haben und einen Mix aus väterlichen und mütterlichen Genen darstellen.
Das Ergebnis sind also jeweils unterschiedliche Keimzellen. Wenn sich somatische Zellen reproduzieren, teilen sie sich im Rahmen der Mitose:
Das Ergebnis sind zwei identische Tochterzellen. Zur Vorbereitung auf
die nächstfolgende Mitose werden die Chromatiden während der
sogenannten Synthesephase redupliziert.
Ein beträchtlicher Teil der Zellen einer erwachsenen Person befinden sich in ihrer G0-Phase
(s. unten), in der sie die Mitoseaktivität eingestellt haben. Das
trifft z.B. auf Nerven- und Muskelzellen zu; diese sind sehr
spezialisiert und erneuern sich kaum mehr. Andere teilen sich täglich,
wie z.B. Epithelzellen im Gastrointestinaltrakt.
Die im Zellkern gespeicherte Erbinformation dient - je nach Abschnitt des Zellzyklus, der normalerweise aus vier Phasen besteht: G1, S, G2 und M -
 der “Arbeitsfunktion” (Interphase),
in der Gene exprimiert (abgelesen und "übersetzt") werden (Synthese von RNA und Proteinen - die
Chromatinfäden liegen im Zellkern als mehr oder weniger entfaltete, gut
ablesbare Fäden
vor) und (vor der nächsten Mitose) die Chromatiden repliziert werden (was
zur Bildung von Schwesterchromatidenpaaren führt -
Replikationsgeschwindigkeit: Bei Bakterien bis zu ~1000, in
eukaryotischen Zellen ~50 Nukleotide pro Sekunde), oder
der “Arbeitsfunktion” (Interphase),
in der Gene exprimiert (abgelesen und "übersetzt") werden (Synthese von RNA und Proteinen - die
Chromatinfäden liegen im Zellkern als mehr oder weniger entfaltete, gut
ablesbare Fäden
vor) und (vor der nächsten Mitose) die Chromatiden repliziert werden (was
zur Bildung von Schwesterchromatidenpaaren führt -
Replikationsgeschwindigkeit: Bei Bakterien bis zu ~1000, in
eukaryotischen Zellen ~50 Nukleotide pro Sekunde), oder
 der Bildung neuer Zellen (Kondensation der Chromatiden zu Chromosomen, Voraussetzung für die Mitose).
der Bildung neuer Zellen (Kondensation der Chromatiden zu Chromosomen, Voraussetzung für die Mitose).
Phase
|
Funktion
|
G1 (growth)
|
Vorbereitung auf DNA-Synthese
|
S (synthesis)
|
DNA-Synthese
Duplikation der Chromosomen
|
G2 (growth)
|
Vorbereitung auf Zellteilung
|
M (mitosis)
|
Teilung in Tochterzellen
|
Die Interphase - in der das Wachstum von Zellen, die sich kontinuierlich teilen, andauert - wird unterteilt ( Abbildung) in die G1, S- und G2-Phase:
Abbildung) in die G1, S- und G2-Phase:
 G1-Phase
(Gap 1): Dies ist der "Arbeitszustand", in dem die (diploide) Zelle ihre
Dienste für den Organismus leistet. Ihre Mindestdauer beträgt ~10
Stunden (die Zelle wächst hier auch und kann sich nicht sofort wieder
teilen). Die Dauer hängt von den Bedingungen ab, in denen sich die Zelle wiederfindet. Signale durch Mitogene
heben die Wirkung intrazellulär hemmender Kontrollfaktoren auf; die
Zelle muss aber auch entscheiden, ob genügend Ressourcen für weitere
Mitosen zur Verfügung stehen - unabhängig von der Anwesenheit mitogener
Signale.
G1-Phase
(Gap 1): Dies ist der "Arbeitszustand", in dem die (diploide) Zelle ihre
Dienste für den Organismus leistet. Ihre Mindestdauer beträgt ~10
Stunden (die Zelle wächst hier auch und kann sich nicht sofort wieder
teilen). Die Dauer hängt von den Bedingungen ab, in denen sich die Zelle wiederfindet. Signale durch Mitogene
heben die Wirkung intrazellulär hemmender Kontrollfaktoren auf; die
Zelle muss aber auch entscheiden, ob genügend Ressourcen für weitere
Mitosen zur Verfügung stehen - unabhängig von der Anwesenheit mitogener
Signale.

 Abbildung: Phasen des Zellzyklus
Nach einer Vorlage in S.M. Liu @ www.ch.ic.ac.uk
Abbildung: Phasen des Zellzyklus
Nach einer Vorlage in S.M. Liu @ www.ch.ic.ac.uk
G steht für gap, Abstand, Lücke - zwischen S- und M-Phasen).
G1- und S- (Synthese-) Phasen
nehmen jeweils ~40% der Dauer eines Zellzyklus in Anspruch (Mensch: im
Schnitt mindestens ~10 und 6-7 Stunden), die G2-Phase ~20% (Mensch: ~4 Stunden); die M- (Mitose-) Phase nur wenige % (~1 Stunde).
Die Länge der Ruhephase ist variabel


Zahlreiche Zellen treten
postmitotisch in die
G0-Phase ein (diese wird auch als
Ruhephase
(resting stage) bezeichnet - was sich auf die Mitoseaktivität bezieht, die Zellen selbst können in der G
0-Phase
hochaktiv sein, z.B. wachsen, sich bewegen, migrieren, Stoffe aufbauen
etc). In diesem modifizierten G
1-Stadium verharren sie entweder

vorübergehend (wenn auch u.U. sehr lange,
z.B. Stammzellen). Aus dieser Phase können (bei G
1)
Zellen in den Zyklus jederzeit wieder eintreten, wenn sie entsprechend
stimuliert werden (z.B. durch Wachstumsfaktoren). Für gealterte Zellen
gilt das nicht. Auch vollständig ausdifferenzierte Zellen verbleiben
normalerweise in G
0;

oder permanent (das trifft auf die meisten Nervenzellen sowie auf das Myokard zu,
die
dauerhaft spezialisiert bleiben).
Auch viele Hepatozyten (Leberparenchymzellen) verharren in der G
0-Phase, können aber (insbesondere bei Gewebeschäden) rasch wieder in die Mitosephase eintreten
(regeneratives Wachstum) und allfälligen Zellverlust wieder wettmachen (nach Entfernung
eines Teils des Organs kann verbliebenes Lebergewebe die Lücke wieder
füllen).

In der G
1- und G
0-Phase vollbringt das Genom den Großteil seiner Arbeit (
Transkription).
Das "Erwecken" der Zellen zu weiteren Teilungen wird (unter günstigen Bedingungen, z.B. ausreichend Nährstoffe) durch
Mitogene verursacht. Diese bremsen - über Wirkung an
Mitogenrezeptoren und z.B. über
Ras (eine GTPase) und
MAP-Kinasen
- die Aktivität cyclinabhängiger
Kinasen (Cdks, s. unten) und triggern
damit auf komplexe Weise den Wiedereintritt der Zelle in den
Teilungszyklus. Voraussetzung für einen Wiedereintritt in den
Zellzyklus ist aber auch ausreichende Verfügbarkeit von Nährstoffen,
Sauerstoff u.a.
Am Ende der G
1-Phase steht die Duplikation der
Zentrosomen (in denen jeweils ein Zentriolenpaar eingeschlossen ist, an dem Mikrotubuli entspringen) und der
G1-Restriktionspunkt, der die Teilungsrate kontrolliert, und danach der
G1/S-Kontrollpunkt (Checkpoint), an dem die Integrität der DNA überprüft wird (s. unten).

In der
S-Phase
(DNA-
Synthesephase: Reduplikation) kopiert (verdoppelt) die Zelle ihr Erbmaterial
(
DNA-Reduplikation).
In der
frühen S-Phase befindet sich in jedem Chromosom
eine DNA-Doppelhelix (Chromosomen 2N, DNA 2C), in der
späten S-Phase - nach der Reduplikation - verfügt jedes Chromosom über
zwei DNA-Doppelhelices (Chromosomen 2N, DNA 4C - vgl.
dort). Die beiden Tochterzellen verfügen nach der Zellteilung wieder über 2N / 2C.
Di S-Phase dauert 6-12 Stunden (etwa 3 Milliarden
Basenpaare werden in dieser Zeit kopiert) und benötigt etwa die Hälfte
der Zeit, die der gesamte Zellzyklus beansprucht. Die Verdoppelung der DNA beginnt jeweils an bestimmten Stellen, den
Replikationsursprüngen (von diesen gibt es pro Chromosom zahlreiche - beim Menschen pro Zelle insgesamt etwa 3-5.10
4 -, was die Replikation - die sonst mehr als einen Monat dauern würde - entsprechend beschleunigt).
In der frühen G1-Phase (bei niedriger CDK-Aktivität, s. unten) bildet sich ein aus Initiatorproteinen bestehender
Prä-Replikationskomplex (
pre-replication complex
pre-RC) an der Anfangsstelle der Replikation (mindestens eine pro
Chromosom). Dabei wird sichergestellt, dass die Replikation
vollständig, aber pro Zellzyklus nur einmal erfolgt. Weiters bauen
SMC-Proteine
(structural maintenance of chromosomes) - zusammen mit Nicht-SMC-Proteinen -
SMC-Komplexe auf, welche die Chromatiden neu gebildeter Chromosomen zusammenhalten (daher die Bezeichnung
Cohesine
für einige dieser Proteinkomplexe). Mikrotubuli der Mitosespindeln
befestigen sich über Cohesine an einem Proteinkomplex namens
Kinetochor 
(das innere Kinetochor ist fest an die DNA befestigt, das äußere interagiert mit Mikrotubuli des Spindelapparates).
Am Ende dieser Phase
bestehen die Chromosomen aus zwei identischen Chromatiden, die Zelle
ist
tetraploid.
 Die beiden Schwesterchromatiden bleiben dann eng aneinandergeheftet und
werden zur Bildung von Mitosechromosomen weiter kondensiert.
Die beiden Schwesterchromatiden bleiben dann eng aneinandergeheftet und
werden zur Bildung von Mitosechromosomen weiter kondensiert.
In der S-Phase nimmt die Synthese von Proteinen zu,
die zusammen mit der DNA Chromatiden aufbauen (z.B. Histonproteine zum
Aufbau neuer Nukleosomen).
Kinetochore sind paarweise um das (die Chromosomenarme "zusammenhaltende")
Zentromer
angeordnet und steuern den Auf- und Abbau der Mikrotubuli und damit die
mitotische Wanderung der Chromosomen. Um die außen von Spindelpol zu
Spindelpol laufenden
polaren Mikrotubuli erstrecken sich
Kinetochor-Mikrotubuli,
die jeweils ein Chromosom mit den beiden Spindelpolen verbinen und die
beiden Chromatiden in der Anaphase zum Spindelpol ziehen.
 G2-Phase (Gap 2)
G2-Phase (Gap 2) dient der finalen Vorbereitung auf die Mitose. In dieser
~4 Stunden dauernden Zeit der "
Kontemplation" kontrolliert die Zelle das Ergebnis der
vorausgegangenen Reduplikationsvorgänge (DNA korrekt verdoppelt?). Am abschließenden
G2/M-Checkpoint (G
2/M-Übergang) werden eventuell notwendige
Korrekturen
an der replizierten DNA vorgenommen (gesamte DNA verdoppelt?). Fällt
die Überprüfung zufriedenstellend aus, erfolgt der Eintritt in die
Mitose; sind bei der Replikation
zu viele Fehler passiert, wird

der Vorgang verzögert, um die entsprechenden Reparaturen vorzunehmen,

die Zelle mitotisch stillgestellt ("Seneszenz", vor allem durch Wirkung des
P53-Proteins, s. unten), oder

die Zelle der
Apoptose zugeführt.
 Details zum Ablauf der Mitose s. weiter unten
Details zum Ablauf der Mitose s. weiter unten


Abbildung: Kontrollpunkte (Restriktionspunkte) des Zellzyklus
Nach einer Vorlage in Campbell / Urry /Cain / Wasserman / Minorsky / Orr: Biology - A Global Approach (12th ed.). Pearson Education 2021
An
"Kontrollpunkten" (durch rote Wände dargestellt) überprüft die Zelle
das Resultat vorausgegangener Vorgänge im Rahmen des Zellzyklus:
Am G1-Kontrollpunkt erfolgt Prüfung auf Nährstoffverfügbarkeit, Zellgröße, Wachstumsfaktoren, DNA-Schäden
Am G2-Kontrollpunkt erfolgt Prüfung auf erfolgreiche DNA-Replikation
Am M-Kontrollpunkt (M = Mitose) erfolgt Prüfung auf die Anheftung von Chromosomen an Spindelfasern

Die Zelle überprüft den Status der Interphase - und damit die Resultate der vorangenangenen Vorgänge - an definierten Kontrollpunkten (checkpoints, restriction points), Stationen zwischen den Phasen des Zellzyklus ( Abbildung): G1/S, G2/M,
und ein Metaphase- zu- Anaphase- Kontrollpunkt. An diesen
Übergangsstellen können allfällig notwendige Reparaturen vorgenommen
werden.
Abbildung): G1/S, G2/M,
und ein Metaphase- zu- Anaphase- Kontrollpunkt. An diesen
Übergangsstellen können allfällig notwendige Reparaturen vorgenommen
werden.
 Kontrollpunkt 1. Am G1-Restriktionspunkt
(G1/S-Übergang) fällt die Entscheidung, ob die Zelle in die Synthesephase eintreten kann. Dieser Punkt ist komplex reguliert, er liegt an einer besonders entscheidenden Stelle (ist die
Zelle zur Reproduktion befähigt? Liegen Signale vor, welche die Mitose begünstigen?). Lautet die Antwort "ja", sind die weiteren Vorgänge
von Außenfaktoren unabhängig, und es erfolgt die DNA-Reduplikation. Dazu ist z.B. die Anwesenheit
bestimmter extrazellulärer Signale erforderlich (Wachstums- und
Adhäsionsfaktoren). Wachstumsfaktoren müssen an ihre Rezeptoren binden, Proteine in der Zelle phosphoryliert
werden und Proliferationsvorgänge starten.
Kontrollpunkt 1. Am G1-Restriktionspunkt
(G1/S-Übergang) fällt die Entscheidung, ob die Zelle in die Synthesephase eintreten kann. Dieser Punkt ist komplex reguliert, er liegt an einer besonders entscheidenden Stelle (ist die
Zelle zur Reproduktion befähigt? Liegen Signale vor, welche die Mitose begünstigen?). Lautet die Antwort "ja", sind die weiteren Vorgänge
von Außenfaktoren unabhängig, und es erfolgt die DNA-Reduplikation. Dazu ist z.B. die Anwesenheit
bestimmter extrazellulärer Signale erforderlich (Wachstums- und
Adhäsionsfaktoren). Wachstumsfaktoren müssen an ihre Rezeptoren binden, Proteine in der Zelle phosphoryliert
werden und Proliferationsvorgänge starten.
Cycline sind Proteine, deren Konzentration zellzyklusunabhängig
ist. Sie haben selbst keine enzymatische Aktivität, sondern binden an
Mitglieder einer Proteinkinase-Familie, die als cyclinabhängige Kinasen
(CDK, cyclin-dependent kinases)
bezeichnet werden. Diese werden dadurch aktiviert und phosphorylieren
für das Fortschreiten des Zellzyklus entscheidende Enzyme
( Abbildung):
Abbildung):

 Abbildung: Aktivierung einer cyclinabhängigen Kinase (cdk)
Abbildung: Aktivierung einer cyclinabhängigen Kinase (cdk)
Nach einer Vorlage in Ritter / Flower / Henderson / Loke / MacEwan / Rang, Rang & Dale's Pharmacology, 9th ed. Elsevier 2020
Die Kontrollpunkte des Zellzyklus werden durch spezifische Cdk's (cyclin dependent kinases)
reguliert. Cdk's sind an Cycline gebunden, die zu bestimmten Zeiten des
Zellzyklus auf- bzw. abgebaut werden.Das begrenzt die Verfügbarkeit
jedes Cyclin-Cdk-Komplexes.
A: Inaktives Enzym
B: Cdk bindet an
Cyclin und wird aktiviert, wodurch es ein spezifisches Proteinsubstrat
(grün, z.B. ein Enzym) phosphorylieren kann. Cdk's (deren Konzentration
im gesamten Zellzyklus ziemlich konstant bleibt) sind nur in Verbindung
mit ihrem Cyclin aktiv
C: Nach der Phosphorylierung des Substratmoleküls wird Cyclin abgebaut, das verbleibende Cdk verliert seine Aktivität

Cyclinabhängige
Kinasen
(CDK bzw. cdk) liegen in der Zelle in inaktiver Form vor und müssen an
Cyclin koppeln, um ihr Zielprotein phosphorylieren zu können.
Sie steuern zahlreiche Vorgänge im Zellzyklus, wie die Duplikation von
Zentrosomen oder die Mitose selbst. CDK-Cyclin-Komplexe wirken über Phosphatasen
- z.B. zum Abbau der Kernmembran, die Kondensierung von Chromosomen,
oder die Bildung des Spindelapparates. Angriffspunkt der
Phosphorylierung sind dabei Serin- und Threoninreste in
Proteinmolekülen, daher spricht man auch von Serin-Threonin-Kinasen.
Begrenzte Wirkdauer: Wachstumsfaktoren regen sowohl die Produktion von Cyclinen als auch von
cyclinabhängigen Kinasen an. Nach jedem Phosphorylierungsvorgang
wird das involvierte Cyclin durch das Ubiquitin-Proteasom-System
zerstückelt (nachdem es durch enzymatisches Anfügen mehrerer kleiner
Moleküle für den Abbau "markiert" wurde). Dadurch ist die Aktivität des
Enzymkomplexes und seine Wirkung auf den Zellzyklus zeitlich limitiert.
Man unterscheidet acht Gruppen unterschiedlicher Cycline (für die
Kontrolle des Zellzyklus die Cycline A, B, D und E), je nachdem, in
welcher Phase sie aktiv sind. CDKs binden jeweils an eines von vier
Cyclinen. Cyclin A aktiviert CDK 1 und 2, Cyclin B CDK 1, Cyclin D CDK
4 und 6, Cyclin E CDK 2.
 Cyclin D-CDK4 / CDK6 steuert Vorgänge der frühen G1-Phase
Cyclin D-CDK4 / CDK6 steuert Vorgänge der frühen G1-Phase
 Cyclin D/E - CDK2 triggert die S-Phase
Cyclin D/E - CDK2 triggert die S-Phase
 Cyclin A - CDK1 / CDK2 kümmert sich um den Abschluss der S-Phase
Cyclin A - CDK1 / CDK2 kümmert sich um den Abschluss der S-Phase
 Für die Mitose ist Cyclin B - CDK1 essentiell
Für die Mitose ist Cyclin B - CDK1 essentiell
 Die Interphase ist Cyclin A - CDK1 / CDK2 -gesteuert
Eine präzise zeitliche
Abfolge garantiert den korrekten Ablauf des Zellzyklus.
Andererseits gibt es CDK-Inhibitoren (CKIs),
diese wiederum werden von einigen Wachstumsfaktoren ausgeschaltet (und
das Fortschreiten im Zellzyklus so freigegeben -
Die Interphase ist Cyclin A - CDK1 / CDK2 -gesteuert
Eine präzise zeitliche
Abfolge garantiert den korrekten Ablauf des Zellzyklus.
Andererseits gibt es CDK-Inhibitoren (CKIs),
diese wiederum werden von einigen Wachstumsfaktoren ausgeschaltet (und
das Fortschreiten im Zellzyklus so freigegeben -  Abbildung).
Abbildung).

 Abbildung: Zellzyklus und Rolle von cdk-Komplexen
Abbildung: Zellzyklus und Rolle von cdk-Komplexen
Nach einer Vorlage in Ritter / Flower / Henderson / Loke / MacEwan / Rang, Rang & Dale's Pharmacology, 9th ed. Elsevier 2020
Der
Ablauf des Zellzyklus ist durch sequentielle Aktivität von Cyclin-cdk-
Komplexen (farbige Pfeile: Cycline D, E, A und B mit zugehörigen cdk's)
gesteuert.
Die Dicke der Pfeile deutet die jeweilige Stärke der cdk-Aktivität im Zellzyklus an. Diese wird durch cdk-Inhibitoren reguliert.
Der chromosomale Status ist angedeutet - haploid in G1, diploid in G2, während der Mitose (M)

Kontrollfaktoren. Das p53-Gen (Tumorsuppressorgen, 'guardian of the genome') codiert für das p53-Protein, das den Zellzyklus am Kontrollpunkt 1 (vor dem Eintreten in die S-Phase) anhalten kann.
Dieses kurzlebige Protein (Halbwertszeit <20 Minuten) ist in der
Zelle meist nur in sehr geringer Menge vorhanden. Treten DNA-Schäden auf,
steigt seine Konzentration
in der Zelle stark an. p53 verhindert dann die weitere Teilung der
Zelle (es "schaltet auf rot") und ermöglicht der Zelle, die beschädigte
DNA zu reparieren. Schlagt dies fehl, kann p53 die Apoptose der Zelle
einleiten.
Auch aktiviert p53 die Transkription des Gens für Protein p21. Dieses
Protein ist besonders bedeutsam; es wird exprimiert, wenn DNA-Schäden
eine Ablesung des p53-Gens auslösen. Das führt zum Anhalten des
Zellzyklus am G1/S-Restriktionspunkt: p21 inaktiviert Cyclin-cdk-Komplexe und verhindert so die Phosphorylierung - und führt zu Unterphosphorylierung - des Rb-Proteins. Rb ist ein zentraler Faktor für die Kontrolle des G1-Restriktionspunkts.
Rb-Protein (in Retinoblastomen entdeckt, daher die Bezeichnung) ist multifunktional (bindet an mindestens 100 andere Proteine). Es liegt in der Zelle in
ziemlich konstanter Konzentration vor; seine Aktivität wird über seine
Phosphorylierung gesteuert. Es inaktiviert in ruhenden Zellen einen Transkriptionsfaktor (E2-Faktor, E2 F). Wird Rb phosphoryliert, lässt es E2 F frei, dieses triggert die Aktivierung von S-Phase-Genen und
die DNA-Replikation kann beginnen.
Unterphosphorylierung
des Rb-Proteins bewirkt ein Anhalten des Zellzyklus am Kontrollpunkt 1. So besteht die Möglichkeit,
die DNA zu reparieren. Ist dies erfolgt, wird der Zyklus fortgesetzt;
wenn nicht, triggert das p53-Gen den Zelluntergang. Dadurch werden
Tochterzellen - und auch Folgegenerationen - vor einer Weitergabe genetischer Schäden bewahrt.
Kontrollproteine sind bedeutsame Wächter über die Intaktheit des Zellzyklus. Da sie bei Tumoren verändert gefunden werden (bei allen Tumorarten ist Rb irgendwie beeinflusst), werden sie als Tumorsuppressoren
bezeichnet. Sie alle sind darauf abgestellt, das Wachstum von Zellen im
Zaum zu halten. Andererseits gibt es auch fördernde Faktoren; Gene mit
dem Bauplan solcher Proteine bezeichnet man als Protoonkogene.
 Onkogene
sind Gene, die meist Wachstumsfaktoren, deren Rezeptoren,
Signaltransducer oder Transkriptionsfaktoren codieren. Mutieren sie,
werden sie oft überaktiv oder resistent gegenüber Hemmsignalen; das
kann Krebsbildung zur Folge haben. Die meisten Onkogene beginnen diese
Entwicklung als Protoonkogene;
diese unterstützen das Zellwachstum und beteiligen sich an der Hemmung einer
Apoptose. Mutieren Protoonkogene unter Viren-,
Strahlungs-, oder Mutageneinfluss, kann sich ihre Funktion verstärken (gain-of-function mutation). Dies kann zu Tumorentstehung führen; die Mutation eines einzigen Gens reicht schon dazu aus.
Onkogene
sind Gene, die meist Wachstumsfaktoren, deren Rezeptoren,
Signaltransducer oder Transkriptionsfaktoren codieren. Mutieren sie,
werden sie oft überaktiv oder resistent gegenüber Hemmsignalen; das
kann Krebsbildung zur Folge haben. Die meisten Onkogene beginnen diese
Entwicklung als Protoonkogene;
diese unterstützen das Zellwachstum und beteiligen sich an der Hemmung einer
Apoptose. Mutieren Protoonkogene unter Viren-,
Strahlungs-, oder Mutageneinfluss, kann sich ihre Funktion verstärken (gain-of-function mutation). Dies kann zu Tumorentstehung führen; die Mutation eines einzigen Gens reicht schon dazu aus.
 Kontrollpunkt 2 (G2/M-Übergang). Der G2/M-Kontrollpunkt ist weniger komplex reguliert als G1/S und stellt eine Art Notbremse
dar, etwa für den Fall, dass die Replikation fehlerhaft verlaufen ist (s. oben). Die Aktivität der Kinasen schwankt mit dem Zellzyklus. So ist am G2/M-Übergang
die cdk-Aktivität hoch, und Proteine, welche die
Vorgänge der frühen Mitose steuern, werden vermehrt aktiviert (Abbau der Kernhülle, Kondensation
der Chromosomen, Aufbau der Spindeltubuli).
Kontrollpunkt 2 (G2/M-Übergang). Der G2/M-Kontrollpunkt ist weniger komplex reguliert als G1/S und stellt eine Art Notbremse
dar, etwa für den Fall, dass die Replikation fehlerhaft verlaufen ist (s. oben). Die Aktivität der Kinasen schwankt mit dem Zellzyklus. So ist am G2/M-Übergang
die cdk-Aktivität hoch, und Proteine, welche die
Vorgänge der frühen Mitose steuern, werden vermehrt aktiviert (Abbau der Kernhülle, Kondensation
der Chromosomen, Aufbau der Spindeltubuli).
Am Metaphase- zu- Anaphase- Kontrollpunkt wird die Trennung der Schwesterchromatiden überprüft; am Ende steht der Abschluss von Mitose und Zytokinese.
 Als Mitose bezeichnet man die Aufteilung des Chromosomensatzes auf zwei Tochterkerne, als Zytokinese die Aufteilung des Zytoplasmas auf zwei Tochterzellen.
Als Mitose bezeichnet man die Aufteilung des Chromosomensatzes auf zwei Tochterkerne, als Zytokinese die Aufteilung des Zytoplasmas auf zwei Tochterzellen.
Die Dauer des gesamten Zellzyklus hängt von
Spezies und Zelltyp ab; der vollständige Durchlauf benötigt bei menschlichen Zellen in vivo mindestens einen Tag. In vitro (Zellkultur) beobachtet man Zykluszeiten
zwischen 12 und 24 Stunden (bei humanen Zelllinien im Schnitt knapp 20
Stunden).
 Differenzierung: Manche (ausdifferenzierte) Zellen, wie Nerven- oder Muskelzellen, teilen sich im Körper gar nicht mehr; sie verlängern ihre G1-Phase - die dann als G0-Phase bezeichnet wird - auf unbestimmte Zeit. Eine Rückkehr in die G1-Phase
ist bei Bedarf möglich (z.B. Lymphozyten, Hepatozyten); wenn sich die
Zelle endgültig differenziert, gibt es aber kein Zurück zum Teilungszyklus
mehr. Wie sich die Zelle entscheidet, hängt von ihrer Umgebung ab (Anwesenheit von Adhäsionsmolekülen / Wachstumsfaktoren).
Differenzierung: Manche (ausdifferenzierte) Zellen, wie Nerven- oder Muskelzellen, teilen sich im Körper gar nicht mehr; sie verlängern ihre G1-Phase - die dann als G0-Phase bezeichnet wird - auf unbestimmte Zeit. Eine Rückkehr in die G1-Phase
ist bei Bedarf möglich (z.B. Lymphozyten, Hepatozyten); wenn sich die
Zelle endgültig differenziert, gibt es aber kein Zurück zum Teilungszyklus
mehr. Wie sich die Zelle entscheidet, hängt von ihrer Umgebung ab (Anwesenheit von Adhäsionsmolekülen / Wachstumsfaktoren).
Differenzierung bedeutet also eine Restriktion des Zugangs zu genetischer Information
und damit zur Synthese betreffender Nukleinsäuren und Proteine. Wie
"erinnert" sich die Zelle, welche Gene im Rahmen der Differenzierung
ablesbar bleiben und welche der Transkription nicht mehr zugänglich sind? Die Differenzierung fixiert epigenetisch Modifikationen der Struktur des Chromatins:
 Demethylierung der DNA und Acetylierung von Histonen bewirken eine "offene" Konformation mit Ablesbarkeit betreffender Genorte,
Demethylierung der DNA und Acetylierung von Histonen bewirken eine "offene" Konformation mit Ablesbarkeit betreffender Genorte,
 Methylierung
der DNA und Deacetylierung von Histonen hingegen eine "geschlossene"
Konformation mit behinderter Transkription (Gene "abgeschaltet").
Methylierung
der DNA und Deacetylierung von Histonen hingegen eine "geschlossene"
Konformation mit behinderter Transkription (Gene "abgeschaltet").
So wirken die Methylierungsmuster an den Chromosomen wie "Lesezeichen",
die darüber orientieren, wo eine Transkription erfolgen kann und wo
nicht.
Steuerung der Mitose 
Auf die G2-Phase kann eine Zellteilung folgen. Diese M-Phase besteht aus zwei Teilen: Mitose (Aufteilung des kopierten Chromosomensatzes auf zwei Tochterkerne) und Zytokinese
(Aufteilung des Zytoplasmas auf zwei Tochterzellen). Dieser Vorgang
dauert typischerweise weniger als eine Stunde. Man schätzt, dass im
Laufe des Lebens im Körper eines Menschen etwa 1017 (hundert Billiarden) Mitosen ablaufen.
Regulierung: Somatische Zellteilungen (Mitosen) werden durch chemische Signale (Mitogene) ausgelöst.
 Ein Mitogen
ist eine Substanz - meist ein Protein -, die Zellen zur Teilung anregt.
Dabei werden Wege zur Signaltransduktion stimuliert, in welche mitogenaktivierte Proteinkinasen (MAPK, MAP-Kinasen)
involviert sind. Diese aktivieren die Ablesung von Genen, u.a. für den
Transkriptionsfaktor MYC (ein zentraler Expressionsregulator), der
wiederum zahlreiche weitere Gene - z.B. für Cyclin A - aktiviert, was
schließlich das Fortschreiten der S-Phase vorantreibt.
Ein Mitogen
ist eine Substanz - meist ein Protein -, die Zellen zur Teilung anregt.
Dabei werden Wege zur Signaltransduktion stimuliert, in welche mitogenaktivierte Proteinkinasen (MAPK, MAP-Kinasen)
involviert sind. Diese aktivieren die Ablesung von Genen, u.a. für den
Transkriptionsfaktor MYC (ein zentraler Expressionsregulator), der
wiederum zahlreiche weitere Gene - z.B. für Cyclin A - aktiviert, was
schließlich das Fortschreiten der S-Phase vorantreibt.
 MAP-Kinasen
regulieren neben der Mitosetätigkeit auch Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose der Zelle.
MAP-Kinasen
regulieren neben der Mitosetätigkeit auch Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose der Zelle.
 Mitogene sind extrazelluläre Signalstoffe, die typischerweise
intrazelluläre "Bremser" des ersten Kontrollpunktes (G1) "überstimmen"
und dadurch den Eingang zur Synthesephase der betreffenden Zelle
öffnen. Sie tun dies, indem sie an Tyrosinkinase-Rezeptoren in der
Zellmembran binden und so intrazelluläre Folgeraktionen (über Ras, MAP)
anstoßen, die in der Freigabe der Mitose münden.
Mitogene sind extrazelluläre Signalstoffe, die typischerweise
intrazelluläre "Bremser" des ersten Kontrollpunktes (G1) "überstimmen"
und dadurch den Eingang zur Synthesephase der betreffenden Zelle
öffnen. Sie tun dies, indem sie an Tyrosinkinase-Rezeptoren in der
Zellmembran binden und so intrazelluläre Folgeraktionen (über Ras, MAP)
anstoßen, die in der Freigabe der Mitose münden.

 Abbildung: Mitosezyklus
Abbildung: Mitosezyklus
Nach einer Vorlage in Leeper-Woodford / Adkison, Integrated Systems. Lippincott Illustraded Reviews, Wolters Cluver 2016
Nach
Ablauf der Telophase entstehen zwei Interphasenkerne. Treten diese in
eine nächste Mitose ein, durchläuft die Zelle wieder einen Zyklus, der
als Prophase bis Telophase organisiert ist (Beschreibung im nachstehenden Text).
Die bei der Replikation entstandenen Schwesterchromatiden werden durch
Proteinkomplexe (Cohesine) zusammengehalten (wie genau, ist noch
unklar). Mit Beginn der Anaphase verschwinden die Cohesinbrücken auch
im Bereich der Centromere, die (nunmehr selbständigen) Chromosomen
können sich auseinander bewegen und werden vom Spindelapparat zu
gegenüber liegenden Zellpolen gezogen.
Der Übersichtlichkeit halber ist nicht der gesamte Chromosomensatz, sondern sind nur drei Chromosomen dargestellt

 Bei der Zellteilung entfernen sich Zentriolen voneinander (bedingt durch Mikrotubuli,
die zwischen ihnen entstehen) und beteiligen sich am Aufbau eines
Spindelapparates, der jeweils zwei Chromatidensätze voneinander trennt.
Dabei greifen die Mikrotubuli des Spindelapparates über als Kinetochor
Bei der Zellteilung entfernen sich Zentriolen voneinander (bedingt durch Mikrotubuli,
die zwischen ihnen entstehen) und beteiligen sich am Aufbau eines
Spindelapparates, der jeweils zwei Chromatidensätze voneinander trennt.
Dabei greifen die Mikrotubuli des Spindelapparates über als Kinetochor bezeichnete Proteinkomplexe an Zentromeren
bezeichnete Proteinkomplexe an Zentromeren an, das sind bis zu 106 Nukleotidpaare lange, stark repetitive DNA-Sequenzen der Chromosomen, an denen diese Verankerung stattfinden kann.
an, das sind bis zu 106 Nukleotidpaare lange, stark repetitive DNA-Sequenzen der Chromosomen, an denen diese Verankerung stattfinden kann.
 Das Zentrosom ist ist eine Zellorganelle, welche Mikrotubuli organisiert. Es besteht aus zwei rechtwinklig zueinander angeordneten Zentriolen, umgeben von einem hochstrukturierten Proteinkomplex (microtubule-organizing center MTOC), das den Spindelapparat bzw. Zilien ordnet. Zentromeren sind Chromosomenregionen, die während einer Zellteilung zwei homologe Chromosomen miteinander verbinden. An diesen bilden sich Kinetochoren aus,
das sind Proteinkomplexe, an denen - während Zellteilungen (Mitosen,
Meiosen) - Mikrotubuli der Mitosespindel befestigt sind.
Das Zentrosom ist ist eine Zellorganelle, welche Mikrotubuli organisiert. Es besteht aus zwei rechtwinklig zueinander angeordneten Zentriolen, umgeben von einem hochstrukturierten Proteinkomplex (microtubule-organizing center MTOC), das den Spindelapparat bzw. Zilien ordnet. Zentromeren sind Chromosomenregionen, die während einer Zellteilung zwei homologe Chromosomen miteinander verbinden. An diesen bilden sich Kinetochoren aus,
das sind Proteinkomplexe, an denen - während Zellteilungen (Mitosen,
Meiosen) - Mikrotubuli der Mitosespindel befestigt sind.
Eine Mitose dauert ungefähr eine Stunde (manchmal auch deutlich länger) und durchläuft konsekutiv folgende komplexen Phasen ( Abbildung):
Abbildung):
 Prophase
Prophase:
Während der Prophase replizieren die Chromosomen und werden zu zwei
Chromatiden, die über ein Kinetochor miteinander verknüpft sind. Die
DNA kondensiert zu kompakten, stabförmigen
Schwesterchromatiden, die zunächst verbunden bleiben (Kohäsion). Der
Spindelapparat beginnt sich zu formieren, und Zentrosomen wandern an
entgegengesetzte Zellpole. Diese Phase kann zwischen
einer halben und (seltener) mehreren Stunden dauern.
 Prometaphase
Prometaphase:
Der Zellkern verliert seine Hülle, die mitotische Spindel haftet sich
an die Kinetochoren (in der Abbildung als gelbe Punkte dargestellt) der
Chromosomen. Die Spindel - aufgebaut aus
Mikrotubuli (bestehend aus α- und β-Tubulin) - kann nicht direkt an ein Zentromer binden, sondern benötigt das Kinetochor als "Vermittler".
 Metaphase
Metaphase:
Der an den Zentriolen entspringende Spindelapparat entwickelt sich, und
die Schwesterchromatiden heften sich paarweise an die Mitosespindel und
werden in der Äquatorialebene angeordnet. In diesem Zeitabschnitt sind
die Chromosomen am stärksten kondensiert (lichtmikroskopisch gut sichtbar). Die Metaphase
dauert etwa 10 Minuten.
Nun nimmt die Menge an CDKs in der Zelle ab, ihre Substratproteine dephosphorylieren, die (E3-)
Ubiquitinaktivität nimmt zu, mitotische Cycline werden abgebaut. Das fördert sie Trennung der neuen Chromosomen.
 Anaphase
Anaphase:
In dieser 2 bis 20 Minuten dauernden Phase verschwindet die Schwesterchromatiden-Kohäsion, die Chromatiden
trennen sich mit Hilfe der Spindelfasern und werden zu den Spindelpolen gezogen. In der Mitte der Zelle bildet sich ein
gürtelförmiger kontraktiler Ring aus
Aktinfilamenten, der die Trennung in zwei Zellen unterstützt (Zytokinese). Die Kohäsion der Chromatiden wird in der Anaphase durch
anaphase-promoting complexes
(APCs) aufgelöst und die Trennung der Chromatiden ermöglicht. Ein APC
besteht aus mehreren Untereinheiten, die z.T. Ubiquitinaktivität haben
und so u.a. Cycline abbaut.
 Telophase
Telophase:
Die Spindel zerfällt, Kernhülle und neue Nukleoli werden
wiederhergestellt, die
Chromosomen in neu gebildete Zellkerne eingefügt und dekondensiert
(entspiralisiert), womit sie wieder in den "Arbeitszustand"
treten können (Ablesung der Erbinformation, die während der Mitose sehr
eingeschränkt ist). Mit der Wiederherstellung der Kernmembran ist die
Telophase abgeschlossen.


Die Interphase besteht aus
- G
1 (gap 1): Synthese von Proteinen, die zur Replikation benötigt werden
- S
(synthesis): Synthese von DNA, RNA und Proteinen
- G
2 (gap 2): Synthese setzt sich fort, DNA wird auf Korrektheit und Stabilität überprüft

Während der M-Phase
(mitosis) teilt sich die Zelle

In der G
0-Phase hat die Zelle den Teilungszyklus verlassen ("Ruhepause" / endgültige Differenzierung / Alterung)

Zu
meiotischen Zellteilungen (Reifeteilung) s.
dort
Genetik beschäftigt sich mit der Biologie der Vererbung. Deren molekulare Grundlage sind die DNA und deren Begleitproteine.
Variationen der Basensequenz in der DNA sind genetischer, andere
Veränderungen (z.B. Methylierungsmuster) epigenetischer Natur, beide
können vererbt werden. Als Genomik bezeichnet man den Teil der Genetik, der sich mit Genen und deren Wechselwirkungen beschäftigt. Als Gen gilt ein DNA-Abschnitt, der zu mRNA umgeschrieben werden kann.
 Das Genom ist die vollständige DNA-Sequenz eines Organismus (bei Viren auch RNA). Das Genom des Menschen ist erstaunlich klein: Es entspricht etwa 750 Megabyte (3.109 DNA-Nukleotide mit jeweils 4 möglichen Basen pro Nukleotid). Die Gesamtzahl der Gene (inklusive Pseudogene) liegt bei ~54.103 (zum Vergleich: E. coli ~4,5.103,
Taufliege ~13,5.103, Wasserfloh ~31.103, Gemüsekohl ~100.103 -
Genomgröße
und Komplexität des Organismus sind kaum korreliert).
Das Genom ist die vollständige DNA-Sequenz eines Organismus (bei Viren auch RNA). Das Genom des Menschen ist erstaunlich klein: Es entspricht etwa 750 Megabyte (3.109 DNA-Nukleotide mit jeweils 4 möglichen Basen pro Nukleotid). Die Gesamtzahl der Gene (inklusive Pseudogene) liegt bei ~54.103 (zum Vergleich: E. coli ~4,5.103,
Taufliege ~13,5.103, Wasserfloh ~31.103, Gemüsekohl ~100.103 -
Genomgröße
und Komplexität des Organismus sind kaum korreliert).
Die Gesamtzahl funktionsfähiger Gene beim Menschen beträgt etwas über 21.000 (mehr als 3000 davon sind Gene, die für Transkriptionsfaktoren codieren). Die Zahl proteincodierender Gene beträgt etwa 20.103.
Die Genbibliothek befindet sich bei diploiden Zellen des
Menschen auf insgesamt 25 Arten von DNA-Molekülen: 22 Autosomen (Chromosom 1 bis 22), 2 Heterosomen (XX / XY) und einer mitochondriellen (zirkulären) DNA (mtDNA).
Chromatinfäden (Chromosomen) können in verschiedener Form vorliegen,
entweder locker gepackt und gut transkribierbar, also ablesbar (Euchromatin  ) oder kompakt, dicht gepackt und schwer bis gar nicht ablesbar (silent state, Heterochromatin
) oder kompakt, dicht gepackt und schwer bis gar nicht ablesbar (silent state, Heterochromatin  ).
).

 Abbildung: Heterochromatin und Euchromatin
Abbildung: Heterochromatin und Euchromatin
Nach Kim YZ, Altered histone modifications in gliomas. Brain Tumor Res Treat 2014; 2: 2288-2405
Heterochromatin (links)
weist methylierte DNA und deacetylierte Histone auf, ist dadurch
kondensiert und der Transkription nicht zugängig (geschlossene
Chromatinanordnung).
Euchromatin (rechts)
hat unmethylierte DNA und acetylierte Histone, weist eine
"aufgelockerte" (offene) Konformation auf und ist der Transkription
zugänglich
 Heterochromatin
beinhaltet im "Arbeitszustand" der Zelle (Interphasenkern) etwa 10% des gesamten Genoms und stellt unterschiedliche Formen der
Chromatinverdichtung dar, die sich auch unterschiedlich auf die
Expression von Genen auswirken - im allgemeinen inaktivierend. Konstitutives Heterochromatin ist immer dicht gepackt (vor allem im Bereich der Zentrosomen und Telomere) und enthält wenige Gene, fakultatives Heterochromatin kann dekondensiert werden und ist reich an Genen. Heterochromatin weist stark gebundenes H1-Histon auf, das den DNA-Faden an Nukleosom fixiert.
Heterochromatin
beinhaltet im "Arbeitszustand" der Zelle (Interphasenkern) etwa 10% des gesamten Genoms und stellt unterschiedliche Formen der
Chromatinverdichtung dar, die sich auch unterschiedlich auf die
Expression von Genen auswirken - im allgemeinen inaktivierend. Konstitutives Heterochromatin ist immer dicht gepackt (vor allem im Bereich der Zentrosomen und Telomere) und enthält wenige Gene, fakultatives Heterochromatin kann dekondensiert werden und ist reich an Genen. Heterochromatin weist stark gebundenes H1-Histon auf, das den DNA-Faden an Nukleosom fixiert.
 Euchromatin
hingegen bindet H1-Histon relativ schwach und zeigt stark methylierte
Histone der Nukleosomenkerne, was mit guter Ablesbarkeit der DNA
korreliert. In differenzierten Zellen ist das Euchromatin nicht
durchgehend "gelockert", sondern unterschiedlich stark kondensiert. Das
Muster der Ablesbarkeit von Genen bestimmt, welche Proteine
synthetisiert werden können und spiegelt die Differenzierung der Zelle
(Myozyt? Lymphozyt? Hepatozyt? etc) wider.
Euchromatin
hingegen bindet H1-Histon relativ schwach und zeigt stark methylierte
Histone der Nukleosomenkerne, was mit guter Ablesbarkeit der DNA
korreliert. In differenzierten Zellen ist das Euchromatin nicht
durchgehend "gelockert", sondern unterschiedlich stark kondensiert. Das
Muster der Ablesbarkeit von Genen bestimmt, welche Proteine
synthetisiert werden können und spiegelt die Differenzierung der Zelle
(Myozyt? Lymphozyt? Hepatozyt? etc) wider.
Werden Bereiche des Euchromatins in Heterochromatin umgewandelt, schalten sich die betreffenden Gene meistens ab (gene silencing).
Solche Vorgänge spielen z.B. im Rahmen immunologischer Regulation - wie
der Ausbildung funktionaler Antigen-Rezeptorgene - eine Rolle.
Besonders stark kondensiert ist das Chromatin während der Mitose
- hier zeigt sich die dichtest mögliche Packung von Chromosomen: Das
Metaphasenchromosom hat nur ein Zehntausendstel der Länge der
vollständig gestreckten DNA.
Sollen Gene abgelesen werden, müssen sie dem Transkriptionsvorgang
zugänglich gemacht werden (Dekondensierung und Volumenzunahme
betreffender Chromatinschleifen). Der Zellkern besteht aus
verschiedenen Zonen mit unterschiedlicher Intensität der Genablesung;
diese Zonen formieren sich dynamisch je nach Bedarf. Sie sind nicht
durch Lipidmembranen von ihrer Umgebung separiert, sondern bilden
dynamische Kern-Subkompartimente. Zellkerne können gleichzeitig hunderte solcher Reaktionszonen beinhalten. Ein besonderes Beispiel ist der Nukleolus mit Netzwerken aus Proteinen und Ribonukleinsäuren.
Kaum mehr als 1% der DNA des Menschen codiert für Proteine. Ein menschliches Gen hat im Durchschnitt 27.000
Nukleotidpaare (größtes Gen: 2,4 Millionen) und enthält zwischen einem
und mehr als 170 Exons (codierende DNA-Sequenzen; eingeschobene nichtcodierende Sequenzen heißen Introns).
Ein durchschnittliches Gen enthält etwa 10 Exons. Gene sind ablesbar,
wenn ihre DNA dekondensiert ("entwirrt") ist.
Nur ein geringer Anteil aller Gene sind in einer durchschnittlichen Zelle zu einem gegebenen Zeitpunkt aktiv. "Pseudogene" sind
durch Mutationen verändert, nicht proteincodierende DNA-Abschnitte können die Genregulation beeinflussen (Regulator-DNA-Sequenzen).
Individualität und genetische Muster:
Mehr
als 99,5% des Genoms sind bei allen Menschen identisch; der
individuelle Unterschied liegt bei den verbleibenden <0,5%
(entsprechend ~15 Millionen Basenpaaren). Zahlreiche Unterschiede
bestehen auf der epigenetischen Ebene (Methylierungsmuster an Cytosinen
der DNA, Modifikationen an Histonen - Methylierung, Acetylierung,
Phosphorylierung u.a.) - diese sind beeinflusst von Umweltfaktoren,
Ernährung, Training, persönlichen Erfahrungen.
Während
der Ontogenese können Gene verändert, vervielfacht, neu
positioniert werden. So können z.B. nach der ersten Teilung der
befruchteten Eizelle zwei genetisch unterschiedliche Zellen entstehen,
und die unterschiedlichen Genome gehen jeweils auf eine Hälfte des
Organismus über (unterschiedlich auf den Körper verteilt). Spätere
Abweichungen beziehen sich dann z.B. nur noch
auf bestimmte Organe usw; die größte genetische Vielfalt scheint im
Gehirn zu bestehen, wo so gut wie jede Nervenzelle eine von den
Nachbarzellen abweichende
genetische Ausstattung aufweisen kann. Lymphozytenklone haben
voneinander abweichende genetische Ausstattung im Bereich der
Erkennungsstellen ihrer Rezeptoren (somatische Rekombination).

 Abbildung: Epigenetische Veränderung - Beispiel Histonmodifikation
Nach einer Vorlage von Ballermann RE 2012, bei dreamerbiologist.wordpress.com
Abbildung: Epigenetische Veränderung - Beispiel Histonmodifikation
Nach einer Vorlage von Ballermann RE 2012, bei dreamerbiologist.wordpress.com
Gene können durch Modifikation der Histon-Endstücke (z.B. durch Acetylierung) ein- oder ausgeschaltet werden
 Das Epigenom bezieht sich auf
Veränderungen an DNA und Histonproteinen, die nicht die Basensequenz
betreffen und u.a. durch DNA-Methylierung, modifizierte Ablesbarkeit
oder Einfluss auf die Genexpression erklärbar sind.
Das Epigenom bezieht sich auf
Veränderungen an DNA und Histonproteinen, die nicht die Basensequenz
betreffen und u.a. durch DNA-Methylierung, modifizierte Ablesbarkeit
oder Einfluss auf die Genexpression erklärbar sind.
Umgebungsbedingungen können das Epigenom verändern, und veränderte
Epigenome können an Nachfahren weitergegeben werden (epigenetische
Vererbung) - auch wenn es umstritten ist, wie groß die Bedeutung der
Übertragung solcher epigenetischer Veränderungen über Generationen
hinweg ist:
Der Großteil der epigenetischen "Einstellungen" (epigenetic settings) - z.B. durch Methylierung - wird in den Urkeimzellen gelöscht und nicht auf neu differenzierte Keimzellen übertragen. Kurz nach der Befruchtung der Eizelle erfolgt eine weitgehende
Demethylierungswelle, und neue (individualspezifische)
Methylierungsmuster entstehen, die sich auf Tochterzellen des
Organismus übertragen. Allerdings: Diese Demethylierungswelle ist nicht komplett, einige der individuellen Erfahrungen sind auf
Nachfolgegenerationen übertragbar (ohne dass dabei Gene
mutiert sind).
Auch die Inaktivierung eines X-Chromosoms
(die nach Entstehen einer XX-Zygote entweder im Chromosom von der
Mutter oder in demjenigen vom Vater erfolgt) ist ein epigenetischer
Vorgang; im Rahmen der Oogenese wird diese Inaktivierung wieder
rückgängig gemacht (beide X-Chromosomen haben dieselbe Wirkchance). Jede
Zelle, die in der Zygote eine diesbezügliche Entscheidung getroffen hat
(väterliches oder mütterliches X-Chromosom inaktiviert) überträgt
dieses Muster auf den gesamten von ihr abstammenden somatischen
Zellklon. Einige Gene können der Inaktivierung übrigens entgehen.
 Zur X-Chromosom-Inaktivierung s. auch dort
Zur X-Chromosom-Inaktivierung s. auch dort

 Abbildung: Grad der DNA-Methylierung als Funktion der individuellen Entwicklung
Abbildung: Grad der DNA-Methylierung als Funktion der individuellen Entwicklung
Nach einer Vorlage in Strachan / Read, Human Molecular Genetics, 5th ed. 2020 (CRC Press)
Mit der
Gametogenese und frühen embryonalen Entwicklung kommt es zu sehr
deutlichen Veränderungen der DNA-Methylierung - oft auch ausgeprägt
gewebespezifisch

Epigenetische Mechanismen
beeinflussen Genaktivitäten unabhängig von der DNA-Basensequenz, indem
sie Gene ein- oder ausschalten können. Beeinflusst werden können z.B.
 die DNA durch Basen-Methylierung (DNA-Methyltransferasen) - die Base bleibt
erhalten, es handelt sich also um keine Mutation. Die Modifikationen sind reversibel (Demethylierung durch DNA-Demethylasen etc), können aber über längere Zeit erhalten bleiben und bilden so die Basis für ein "Zellgedächtnis". Die Dynamik der Methylierungen ist vermutlich der wichtigste epigenetische
Mechanismus
die DNA durch Basen-Methylierung (DNA-Methyltransferasen) - die Base bleibt
erhalten, es handelt sich also um keine Mutation. Die Modifikationen sind reversibel (Demethylierung durch DNA-Demethylasen etc), können aber über längere Zeit erhalten bleiben und bilden so die Basis für ein "Zellgedächtnis". Die Dynamik der Methylierungen ist vermutlich der wichtigste epigenetische
Mechanismus
 Histon-Endstücke (
Histon-Endstücke ( Abbildung oben) durch Anfügen (Writer - z.B. Acetylierung,
Methylierung oder Phosphorylierung), Entfernen (Eraser, z.B. Demethylierung durch Histon-Demethylasen) oder
"Verwalten" von Modifikationen. Modifikationen des Histonmoleküls
können Genaktivitäten verändern und Proteinkomplexe (Reader, Leser-Proteine)
attrahieren. Haben solche Leserkomplexe an den modifizierten
Histonkomplex angedockt, ziehen sie weitere Proteinkomplexe an,
die enzymatisch aktiv sind oder zusätzliche Bindungsstellen
bereitstellen und so die Aktivität abhängiger Gene verändern
Abbildung oben) durch Anfügen (Writer - z.B. Acetylierung,
Methylierung oder Phosphorylierung), Entfernen (Eraser, z.B. Demethylierung durch Histon-Demethylasen) oder
"Verwalten" von Modifikationen. Modifikationen des Histonmoleküls
können Genaktivitäten verändern und Proteinkomplexe (Reader, Leser-Proteine)
attrahieren. Haben solche Leserkomplexe an den modifizierten
Histonkomplex angedockt, ziehen sie weitere Proteinkomplexe an,
die enzymatisch aktiv sind oder zusätzliche Bindungsstellen
bereitstellen und so die Aktivität abhängiger Gene verändern
 die Zugänglichkeit an die DNA durch strukturelle Veränderungen des Chromatins
die Zugänglichkeit an die DNA durch strukturelle Veränderungen des Chromatins
 die Genexpression, beeinflusst durch Interaktion von Histonen und Proteinen. Genexpression nennt man die Transkription eines Gens zu mRNA, seine Translation zu Protein, oder beides.
die Genexpression, beeinflusst durch Interaktion von Histonen und Proteinen. Genexpression nennt man die Transkription eines Gens zu mRNA, seine Translation zu Protein, oder beides.

 Abbildung: Zellzyklus (vereinfacht)
Nach einer Vorlage in histology.leeds.ac.uk
Abbildung: Zellzyklus (vereinfacht)
Nach einer Vorlage in histology.leeds.ac.uk
Zur besseren Übersichtlichkeit nur ein Chromosom gezeigt. Interphase mit Bildung von Schwesterchromatidenpaaren (oben), Mitose (Mitte), Zellteilung und neue Interphase (unten)

Wenn
sich die Zelle teilt, verdichten sich die Chromatiden zu Chromosomen. Es gibt 22
Paare Autosomen - jeweils ein Chromatid vom Vater und eines von der Mutter ( Abbildung), man spricht von homologen Chromosomen (Homologen). Weiters hat die Frau zwei
X-Chromosomen, der Mann ein X- und ein Y-Chromosom
(Heterosomen,
“Geschlechtschromosomen”). Beim Mann stammt das X-Chromosom von der
Mutter und das Y-Chromosom vom Vater (nichthomologes Chromosomenpaar).
Abbildung), man spricht von homologen Chromosomen (Homologen). Weiters hat die Frau zwei
X-Chromosomen, der Mann ein X- und ein Y-Chromosom
(Heterosomen,
“Geschlechtschromosomen”). Beim Mann stammt das X-Chromosom von der
Mutter und das Y-Chromosom vom Vater (nichthomologes Chromosomenpaar).
Bei der Teilung des Zellkerns
(Mitose) wird die DNA auf zwei gleiche Tochterkerne übertragen
(identische Reduplikation). Vorher haben sich zwei Doppelstränge
gebildet, die dann auf die Tochterkerne aufgeteilt werden.
 Über Meiose s. dort
Über Meiose s. dort
Proteinsynthese des Organismus: Insgesamt stellt eine erwachsene Person pro Tag
~400 Gramm Eiweiß neu her. Mehr als 50% des menschlichen Genoms sind repetitive Sequenzen unbekannter Funktion.
Ein weiterer Teil des Genoms codiert kurze RNA-Sequenzen (Mikro-RNA, miRNAs). Diese bedeuten keine Proteine, sondern inhibieren (nach entsprechender Modifikation im Zytoplasma) die Genexpression (gene silencing). miRNAs werden - z.B. infolge erhöhter endothelialer Belastung (shear stress) bei körperlicher Arbeit - in den Kreislauf abgegeben (ci-miRNAs, circulation),
sind hier sehr stabil und können als interzelluläre Signalstoffe
physiologische Vorgänge beeinflussen (das erklärt möglicherweise einen
Teil der gesundheitsfördernden Effekte von Muskelaktivität).
Proteinsortierung: Das (an den Ribosomen) neugebildete Protein kann im Zytosol verbleiben (zytosolischer Weg, vor allem zur Bildung von Enzymen) oder - unter Beteiligung des Golgi-Apparates - zum Export aus der Zelle gebracht werden (sekretorischer Weg).
 Die
Frage, wie Proteine in der Zelle für zytosolische oder sekretorische
Verwendung "sortiert" werden, wurde u.a. vom US-amerikanischen
Biochemiker Günter Blobel
aufgeklärt, der dafür 1999 den Nobelpreis für Physiologie oder Medizin
erhielt. Die Sortierung hängt davon ab, ob die mRNA mit einer Signalsequenz (für das endoplasmatische Retikulum) ausgestattet ist oder nicht. Das "Protein Targeting" erfolgt dann über Translokationssequenzen
- diese bestimmen, wohin (in welches zelluläre Kompartiment) das
gebildete Protein genau gelangt (z.B. in den Zellkern, die
Mitochondrien, etc).
Die
Frage, wie Proteine in der Zelle für zytosolische oder sekretorische
Verwendung "sortiert" werden, wurde u.a. vom US-amerikanischen
Biochemiker Günter Blobel
aufgeklärt, der dafür 1999 den Nobelpreis für Physiologie oder Medizin
erhielt. Die Sortierung hängt davon ab, ob die mRNA mit einer Signalsequenz (für das endoplasmatische Retikulum) ausgestattet ist oder nicht. Das "Protein Targeting" erfolgt dann über Translokationssequenzen
- diese bestimmen, wohin (in welches zelluläre Kompartiment) das
gebildete Protein genau gelangt (z.B. in den Zellkern, die
Mitochondrien, etc).
Die Reihenfolge der Basen am DNA-Strang gibt die
Reihenfolge komplementärer Basen am Parallelstrang an, die
Erbinformation ist doppelt (diploid) angelegt. Zur Reduplikation wird der
Doppelstrang aufgetrennt und die freien Basen geben die Matrize zur
Bildung des neuen DNA-Fadens vor. Im “Arbeitszustand” ist die DNA die Matrize zur Bildung von
Ribonukleinsäuren (RNA); diese werden im Nukleolus des Zellkerns
gebildet.
Physiologie des Zellkerns

vgl.
dort
Das
im Zellkern vorhandene Chromatin bildet komplexe Strukturen, die sich
je nach Zustand dynamisch verändern. Sie sind Ausdruck von
Kompartimentierung einerseits, Interaktion zwischen Nukleinsäuren und Begleitproteinen andererseits ( Abbildung):
Abbildung):

 Abbildung: Aufbau und Funktion des Zellkerns
Abbildung: Aufbau und Funktion des Zellkerns
Nach Razin SV, Ulianov SV. Genome-Directed Cell Nucleus Assembly. Biology 2022; 11: 708
Übersicht links oben: Der Zellkern ist von einer zweifachen Doppellipidschicht (Kernmembran, Kernhülle, nuclear envelope)
umgeben. Sie enthält den perinukleären Spalt, der an mehreren Stellen
direkt in das raue endoplasmatische Retikulum übergeht. Nach innen
liegt sie einer Schichte intermediärer Filamente (rot dargestellt) an,
die aus Lamin bestehen (Kernhülle, Lamina). Das Chromatin (CM: chromatin mass,
dunkelgrau dargestellt) bildet die Grundlage für die
Strukturierung des Zellkerns und seines Nukleolus. Es erstreckt sich
zwischen der unter der Kernmembran liegenden Lamina einerseits und dem
Nukleolus andererseits (strichlierter Doppelpfeil). Die jeweilige
Anhaftung erfolgt durch entsprechende Chromatinabschnitte (LADs: lamina-associated domains, NADs: nucleolus-associated domains).
Zwischen Partien mit kondensiertem Chromatin befinden sich Spalträume
(Interchromatinkompartiment), sie enthalten Nukleoplasma (hellgrau
dargestellt), Kernkörperchen (z.B. Speckles) und
Ribonukleoproteinpartikel. Dieses Kompartiment bietet Transportkanäle
zu Kernporen und endoplasmatischem Retikulum.
Der Nukleolus (meist nur einer pro Zellkern) besteht aus einer äußeren granulären Komponente (granular component GC), der dichten fibrillären Komponente (dense fibrillar component DFC) sowie fibrillären Zentren (fibrillar centers
FC). Die Funktion des Nukleolus besteht im Aufbau von Ribosomen, die
dann (via Kernporen) in das endoplasmatische Retikulum verlagert
werden. Zellen mit besonders intensiver Eiweißsynthese (Ribosomen
werden für die Translation benötigt) haben einen besonders großen Nucleolus.
Inset rechts oben:
Die granuläre Komponente (GC) eines Nucleolus enthält ribosomale
Proteine und das multifunktionale Protein Nukleophosmin. Fibrilläre
Zentren (FC) entstehen mit rDNA-Transkriptionsvorgängen, sie bilden
ribosomale Ribonukleinsäuren (rRNA) und sind jeweils umgeben von einer
dichten fibrillären Komponente (DFC); diese ist transkriptorisch aktiv
und enthält das Enzym Fibrillarin, das vermutlich prä-rRNA bearbeitet.
Der nukleoläre
Remodellierungskomplex (NoRC) im perinukleolären Raum inaktiviert
rDNA-Sequenzen im Nucleolus via Enzyme, die Histone modifizieren
(Histon-Deacetylasen) bzw. DNA methylieren (DNA-Methyltransferasen).
Inset rechts unten: Nuclear speckles
("Kernsprenkel") sind Kernkörperchen. Diese bilden sich unter
dem Einfluss von Transkriptionsvorgängen und Interaktion zwischen RNA
und RNA-bindenden Proteinen. Entfernt liegende DNA-Sequenzen können zu
diesen Arealen rekrutiert werden. Neu synthetisierte RNA kann über
Kanäle des Interchromatinkompartiments zu Kernporen und in das
endoplasmatische Retikulum gelangeen. Nuclear speckles enthalten
Spleißfaktoren, hier wird mRNA modifiziert (Entfernung von Introns).
Jeder Zellkern
enthält einige Dutzend speckles; diese bilden sich je nach Erfordernis
und interagieren in dynamischer Weise mit ihrer Nachbarschaft im
Zellkern. So beteiligen sie sich nicht nur an der Transkription,
sondern auch an Veränderungen des Zytoskeletts (Tubulin, Myosine,
Lamine sind Komponenten der Speckles) und können Proteine nach Bedarf
synthetisieren (zwecks Lokalisierung von Chromosomen, Transkription,
Spleißen etc) oder abbauen (sie enthalten Ubiquitine). Speckles verschwinden mit Beginn einer Zellteilung (Prophase) und treten an deren Ende wieder neu auf (Telophase).
Inset links unten:
Ribonukleoproteine sind Komplexe aus Ribonukleinsäure und RNA-bindenden
Proteinen. Sie bilden sich im Rahmen der Gentranskription und
anschließender posttranskriptioneller Modifikationen der mRNA. Sie
verhindern die Faltung der Prä-mRNA in sekundäre Formen (kann durch
Basenpaaring komplementärer Sequenzen auftreten), welche die
Interaktion mit anderen Proteinen behindern könnte, und helfen beim
Transport der fertigen mRNA aus dem Zellkern. Solange
Proteine an den Komplex gebunden sind, ist die mRNA noch nicht fertig
für den Export in das endoplasmatische Retikulum. Nach vollendetem
Splicing bleiben die Proteine an die Introns gebunden und markieren
diese für den Abbau

Der
Zellkern verfügt nicht über die Maschinerie zur Proteinsynthese, daher
müssen nukleäre Proteine aus dem Zytoplasma importiert werden. Proteine
bis zu 40 kDa können durch spezifische Kernporen importiert werden,
größere bedürfen dazu eigener Lokalisationssignale.
Transkription 
 Als Transkription bezeichnet man das Kopieren von Genen durch Bildung entsprechender RNA. Nicht jede Transkription führt auch zu Translation, also der Synthese entsprechender Proteine. Transkription bedeutet, dass entsprechende Gene exprimiert werden (Genexpression). Unter einem Transkriptom versteht man die Gesamtheit aller zu einem bestimmten Zeitpunkt von einer Zelle hergestellten RNS-Moleküle.
Als Transkription bezeichnet man das Kopieren von Genen durch Bildung entsprechender RNA. Nicht jede Transkription führt auch zu Translation, also der Synthese entsprechender Proteine. Transkription bedeutet, dass entsprechende Gene exprimiert werden (Genexpression). Unter einem Transkriptom versteht man die Gesamtheit aller zu einem bestimmten Zeitpunkt von einer Zelle hergestellten RNS-Moleküle.
Die RNA-Synthese schließt
viele Enzyme ein. Es entstehen Bindungskomplexe, die verschiedene
Stellen der DNA umfassen können. Das Wechselspiel von DNA und
Ablesefaktoren entscheidet, welche Eiweiße von der Zelle gebildet
(exprimiert) werden. Dies kann von innerhalb und außerhalb der Zelle beeinflusst
werden, mit zahlreichen Möglichkeiten der Rückkopplung und
Steuerung.


 Abbildung: Transkription (Bildung von mRNA) und Translation (Proteinsynthese)
Nach einer Vorlage bei Thibodeau / Patton, Anatomy & Physiology (6th ed), Mosby Elsevier 2007
Abbildung: Transkription (Bildung von mRNA) und Translation (Proteinsynthese)
Nach einer Vorlage bei Thibodeau / Patton, Anatomy & Physiology (6th ed), Mosby Elsevier 2007
Nach Ablesung des Bauplans im Zellkern durch Übersetzung (Transkription) entsprechender DNA-Sequenzen (Gene) in Boten-RNA (mRNA) und deren Aufbereitung (Splicing) erfolgt die Verlagerung der mRNA (via Kernporen) in das Zytoplasma.
Hier erfolgt die Proteinsynthese (Translation: Übersetzung von der Nucleinsäure- in die
Aminosäuresprache).
Transfer-RNA (tRNA) -Moleküle schaffen Aminosäuren heran, spezifisch
jeweils entsprechend dem (aus drei Nukleotiden bestehenden) Codon. Am
Ribosom werden die Aminosäuren verknüpft und verlassen - nunmehr als
Bestandteil der wachsenden Peptidkette - den Translationsort

Bei der Ablesung (Transkription) der DNA, die nicht der Replikation dient,
entstehen Ribonukleinsäuren
(RNA). Diese sind ähnlich wie DNA-Ketten aufgebaut, sind aber Einzelstränge,
enthalten Ribose als Zuckeranteil, und Uracil (U) anstelle von Thymin.
Gene werden durch RNA-Polymerasen abgelesen. Dazu müssen sie sich an eine direkt vor dem Gen gelegene Promotorsequenz anlagern. Das können sie aber nicht selbständig - sie brauchen dazu Transkriptionsfaktoren. Diese steuern - wie auch Chromatinstruktur, DNA-Methylierung oder Histon-Acetylierung - die Genexpression (s. oben).
Klassischerweise beginnt die Transkription mit der Anlagerung einer RNA-Polymerase an die Promotersequenz der DNA. Diese "verwaltet" das Gen; sie liegt - fast immer direkt - stromaufwärts (upstream) vom Transkriptions-Startpunkt.
An diesem greift die RNA-Polymerase an, und zwar an basalen Promotorelementen, z.B. der - aus Adenin- und Thyminmolekülen aufgebauten - TATA-Box bei regulierbaren Genen (fast alle) oder dem INR-Element (Initiationselement) bei konstitutiven (ständig exprimierbaren) Genen.
Am Transkriptionsstartpunkt
müssen sich bei eukaryotischen Zellen zahlreiche (>100)
Proteine zusammenfinden, um die Transkription zu starten. Dazu muss
u.a. der DNA-Strang "auseinandergezogen" werden, damit eine Sequenz
ablesbar wird. Es bildet sich ein "Transkriptionsauge" (auch Präinitiationskomplex, RNA polymerase II holoenzyme oder basal transcription machinery genannt, s. oben) - auf einer
Seite ein loser DNA-Strang, auf der anderen ein DNA-RNA-Hybrid für die
Transkription, und als "Vorreiter" eine Helikase, welche die beiden DNA-Stränge voneinander trennt.
Bei diesem Vorgang würde sich die DNA
verzwirbeln; Topoisomerasen lösen vorübergehend die DNA-Stränge auf und verbinden sie nachher wieder.
Enhancersequenzen spielen oft mit eine Rolle: Sie binden regulatorische Proteine,
welche (mittels Mediatormolekül) die RNA-Polymeraseaktivität
beeinflussen (  Abbildung). Dadurch
verändern sich Chromatinstruktur, Aktivität der RNA-Polymerase und
Bindung von Transkriptionsfaktoren. Resultat ist die Verstärkung (oder
Abschwächung) der Proteinsynthese.
Abbildung). Dadurch
verändern sich Chromatinstruktur, Aktivität der RNA-Polymerase und
Bindung von Transkriptionsfaktoren. Resultat ist die Verstärkung (oder
Abschwächung) der Proteinsynthese.

 Abbildung: Multiple Genregulation
Nach einer Vorlage bei nature.com/scitable
Abbildung: Multiple Genregulation
Nach einer Vorlage bei nature.com/scitable
Transkriptionsregulatoren können verschiedene, spezifische Bedeutung haben, und Kombinationen davon (hier A, B und C) können wiederum spezifische Mediatorproteine (orange) attrahieren.
Gene können so auf verschiedene Weise aktiviert werden, abhängig von dem Muster der anregenden Faktoren

Es gibt regulatorische Proteine, welche die Transkription mehrerer
Gene beeinflussen. Das ist einer der Mechanismen, wie die Zelle die
Steuerung zahlreicher Gene gleichzeitig koordiniert. Dabei können
unterschiedliche regulatorische Proteine beteiligt sein, und die
Bindungsstellen an der DNA können in beträchtlichem Abstand zueinander
liegen ( Abbildung).
Abbildung).
Darüber hinaus ist die Genexpression meist von einer Kombination kooperierender Regulationsproteine reguliert - dies erhöht die Flexibilität und Anpassungsfähigkeit der Kontrolle der Genexpression.
Ribonukleinsäuren
stehen phylogenetisch wahrscheinlich am Anfang der biologischen Informationscodierung.
Sie sind chemisch nicht so stabil wie DNA, die sich (außer in
RNA-Viren) als primärer Träger der Erbinformation durchgesetzt hat. Man
kann Ribonukleinsäuren nach ihrer Funktion in zwei Gruppen einteilen:
 Codierende RNA, das sind Boten-RNA (messenger RNA, mRNA), das Produkt der Transkription
(Ablesung der DNA im Zellkern oder in Mitochondrien). mRNA dienen als
Informationsträger für die Synthese von Proteinen (Translation im
Zytoplasma oder in Mitochondrien) und sind (je nach zu codierendem Protein) unterschiedlich lang (bis zu mehrere tausend Basenpaare).
Codierende RNA, das sind Boten-RNA (messenger RNA, mRNA), das Produkt der Transkription
(Ablesung der DNA im Zellkern oder in Mitochondrien). mRNA dienen als
Informationsträger für die Synthese von Proteinen (Translation im
Zytoplasma oder in Mitochondrien) und sind (je nach zu codierendem Protein) unterschiedlich lang (bis zu mehrere tausend Basenpaare).
Vorstufen sind pre-mRNA (precursor messenger RNA)
- diese enthalten neben Exons (zur Proteinsynthese als Schablone
benötigten Bauanleitungen) auch Introns (die entfernt werden müssen, um
das betreffende Zielprotein korrekt zu codieren). An diesem Vorgang
sind sRNAs beteiligt (s. unten).
 Nicht-codierende RNA, eine heterogene Gruppe von Ribonukleinsäuren, die nicht als Bauanleitung für Proteine dienen. Nichtcodierende DNA sind u.a.
Nicht-codierende RNA, eine heterogene Gruppe von Ribonukleinsäuren, die nicht als Bauanleitung für Proteine dienen. Nichtcodierende DNA sind u.a.
 Ribosomale (rRNA) als Bestandteile von
Ribosomen
Ribosomale (rRNA) als Bestandteile von
Ribosomen
 Transfer-RNA (tRNA), welche die jeweils erforderlichen Aminosäuren an den
Transkriptionsvorgang anliefern
Transfer-RNA (tRNA), welche die jeweils erforderlichen Aminosäuren an den
Transkriptionsvorgang anliefern
 Kleine (kurze) RNAs (sRNA, small RNA) zur Regulierung spezifischer Zielgene, zum Spleißen von RNA u.a.
Kleine (kurze) RNAs (sRNA, small RNA) zur Regulierung spezifischer Zielgene, zum Spleißen von RNA u.a.
 Mikro-RNAs (miRNA, microRNA) beteiligen sich an der Regulierung von Transkription und Translation
Mikro-RNAs (miRNA, microRNA) beteiligen sich an der Regulierung von Transkription und Translation
 Nichtkodierende lange RNA (lncRNA, long non-coding RNA)
- definiert als nichtcodierende RNA mit über 200 Nukleotiden mit
Funktionen in der Regulierung von Transkription und
posttranskriptionalen Vorgängen.
Nichtkodierende lange RNA (lncRNA, long non-coding RNA)
- definiert als nichtcodierende RNA mit über 200 Nukleotiden mit
Funktionen in der Regulierung von Transkription und
posttranskriptionalen Vorgängen.
Nichtcodierende RNA unterstützen oft die
Expression von Genen, meist in allgemeiner Weise, manchmal auf
bestimmte Zielgene gerichtet. Sie können die Translation auch reprimieren (wie miRNA).
Messenger-RNA (mRNA) und ihre Vorstufen
mRNA repräsentieren
nur 1-5% der RNA in der Zelle, aber >50% der RNA-Neusynthese.
Sie bestehen aus einigen 103-104
Nukleotiden, deren Sequenz exakt komplementär zur Nukleotidsequenz des
kopierten DNA-Strangs ist. Jeweils drei Nukleotide einer reifen mRNA
bilden ein Codon, das eine bestimmte Aminosäure bedeutet. Das Spektrum der in der Zelle vorhandenen mRNA
hängt vom Zelltypus ab und kann sehr unterschiedlich sein. mRNA werden nach ihrer Synthese (Transkription) rasch wieder abgebaut
(kurze Verweildauer).
Entstehung der mRNA: Bis das fertige mRNA-Molekül (mature mRNA) vorliegt, müssen mehrere Vorgänge ablaufen. Die Transkription eines DNA-Abschnitts von der DNA-Vorlage ergibt zunächst ein primäres Transkript (primary transcript), das - wie auch bei rRNA und tRNA - zu fertiger funktionsfähiger RNA umgewandelt werden muss, bevor es zum Einsatz kommt. Vorstufen der reifen mRNA (precursor messenger RNA, pre-mRNA) enthalten nicht nur proteincodierende Basensequenzen (Exons), sondern auch nicht-proteincodierende Sequenzen (Introns). Der Begriff heteronukleäre RNA (hnRNA) wird oft synonym zu pre-mRNA gebraucht - allerdings kann hnRNA nukleäre RNA-Transkripte inkludieren, die in zytoplasmatischer mRNA nicht enthalten sind.
Durch sogenanntes Spleißen (splicing) werden die Introns noch im Zellkern aus der pre-mRNA entfernt. Introns können regulatorische Aufgaben haben oder eine Rolle beim alternativen Spleißen spielen (dabei können auch Exons aus der RNA entfernt werden, nicht nur Introns). Auf diese Weise kann ein und derselbe mRNA-Abschnitt als Vorlage für unterschiedliche Proteinmoleküle verwendet werden, und das Proteom (Gesamtheit aller Proteinarten) eines Individuums ist vielfältiger als sein Genom (Gesamtheit aller Gene).
Das Entfernen nicht benötigter Basen (meist Introns) aus der mRNA erfolgt im Zellkern üblicherweise in Protein-RNA-Komplexen (Spliceosomen).
Dieser Vorgang und das anschließende Spleißen wiederholen sich
mehrfach, bis alle Exons direkt miteinander verknüpft worden sind. Übrig bleibt die "reife" mRNA - eine Abfolge von Exons als Bauanleitung für ein bestimmtes Protein, fertig für den Export durch Kernporen in das Zytosol.
Redigieren (Reifung) von RNA (RNA editing): An der RNA werden mehrere Veränderungen vorgenommen, bevor die (von Introns befreite) mRNA den Zellkern verlassen kann:

 Anbringen
eines methylierten Guanosinmoleküls (5'-Kappe) am 5'-Ende (hilft wahrscheinlich beim Spleißen, beim Transport aus dem Zellkern und beim "Einfädeln" der Translation im Zytoplasma),
Anbringen
eines methylierten Guanosinmoleküls (5'-Kappe) am 5'-Ende (hilft wahrscheinlich beim Spleißen, beim Transport aus dem Zellkern und beim "Einfädeln" der Translation im Zytoplasma),
 Anbringen eines Poly-A-Schwanzes am 3'-Ende (Funktion: RNA-Stabilisierung, Hilfe beim Transport durch den Zellkern),
Anbringen eines Poly-A-Schwanzes am 3'-Ende (Funktion: RNA-Stabilisierung, Hilfe beim Transport durch den Zellkern),
Das fertige Produkt besteht aus folgenden Teilen ( Abbildung):
Abbildung):
 Dem 5'-Pol mit einer - vor dem Abbau durch Nukleasen schützenden - "Kappe" (Cap),
Dem 5'-Pol mit einer - vor dem Abbau durch Nukleasen schützenden - "Kappe" (Cap),
 einer darauf folgenden nicht proteincodierenden Sequenz (5'-UTR: untranslated region) - diese beeinflusst u.a. Stabilität, Export und Effizienz der mRNA,
einer darauf folgenden nicht proteincodierenden Sequenz (5'-UTR: untranslated region) - diese beeinflusst u.a. Stabilität, Export und Effizienz der mRNA,
 dem peptid- bzw. proteincodierenden Leseraster (ORF: Open reading frame) - dieser beginnt mit einem Start-Codon (AUG) und endet mit einem
Stop-Codon (UAA, UAG oder UGA). Die Translation (Proteinsynthese)
beginnt mit dem Start-Codon und endet nach dem Stop-Codon,
dem peptid- bzw. proteincodierenden Leseraster (ORF: Open reading frame) - dieser beginnt mit einem Start-Codon (AUG) und endet mit einem
Stop-Codon (UAA, UAG oder UGA). Die Translation (Proteinsynthese)
beginnt mit dem Start-Codon und endet nach dem Stop-Codon,
 einer nicht proteincodierenden 3'-UTR (three prime untranslated region), welche direkt dem Stop-Codon folgt und auf mehrfache Art und Weise die Genexpression oder die Stabilität des Transkriptionsprodukts beeinflusst, sowie
einer nicht proteincodierenden 3'-UTR (three prime untranslated region), welche direkt dem Stop-Codon folgt und auf mehrfache Art und Weise die Genexpression oder die Stabilität des Transkriptionsprodukts beeinflusst, sowie
 einem durch Polyadenylierung entstandenen Poly-A-Schwanz (Poly (A) tail) am 5'-Ende der mRNA.
einem durch Polyadenylierung entstandenen Poly-A-Schwanz (Poly (A) tail) am 5'-Ende der mRNA.
Als nächstes werden
die mRNA-Moleküle aus dem Zellkern - durch die Kernmembran (Karyolemm)
- in das endoplasmatische Retikulum exportiert. Die aus bis zu ~100
Proteinen bestehenden Kernporen
haben einen Durchmesser von 9 nm und lassen die Diffusion kleinerer
Moleküle (bis 30 kD) zu. Der Molekülaustausch ist allerdings auch durch
verschiedene Transportmechanismen reguliert (das gilt sowohl für den
Import als auch den Export von Molekülen, wie mRNA).
Hat die
mRNA den Zellkern verlassen, befindet sie sich im Zytoplasma und es
stellt sich die Frage, wie sie zum Ort der
nun folgenden Transkription gelangt - denn dieser ist nicht immer in
der unmittelbaren Umgebung des Kerns, sondern kann auch weit (bis zu
einem Meter
- Nervenzellen!) entfernt sein. Die mRNA wird dann aktiv zum
Translationsort
transportiert (s. dort) - denn das geht einfacher
als der Transport zahlreicher Proteinmoleküle in die Peripherie und
bietet die Möglichkeit, einen mRNA-Vorrat anzulegen und erst bei
Bedarf zu nutzen.
Nun kommen tRNA und rRNA ins Spiel (zu Polymerasen  s. oben):
s. oben):
Transfer-RNA (tRNA)
Transfer-RNA (t-RNA) besteht aus rund 80 Nukleotiden und einer Aminosäure, welche einem bestimmten (in der Mitte des kleeblattförmigen Moleküls gelegenen) Anticodon entspricht. Dabei spiegelt diese Basensequenz die entsprechende DNA-Sequenz wider (z.B. für Prolin: DNA: CCG → mRNA: GGC → tRNA: CCG).
Die tRNA agiert als “Dolmetsch”: Sie bringt in einem Ribosom die (dem Anticodon entsprechende, an einem Ende der Nukleotidkette an ein Adenylat gebundene) Aminosäure zu der Peptidkette, die beim Ablesevorgang der durchlaufenden mRNA-Kette entsteht (s. Abbildung oben). tRNA werden nach der Entkoppelung an das Zellplasma zurückgegeben, wo
sie neue Aminosäuren “tanken”.
 Über den Zugriff extrazellulärer Signalfaktoren auf die Transkription s. dort
Über den Zugriff extrazellulärer Signalfaktoren auf die Transkription s. dort

Über
Translation s.
unten
Ribosomale RNA (rRNA) fungiert als Baustein von Ribosomen (Leberzellen
mit ihrem ausgeprägten Proteinstoffwechsel enthalten je ~4 Millionen
davon) und macht etwa 60% ihrer Masse aus (40% liefern etwa 75
verschiedene Proteintypen - Enzyme und Strukturträger). Ribosomen sind
kurzlebige Zellorganellen; sie werden innerhalb von ~6 Stunden ersetzt
(pro Leberzelle etwa 180 pro Sekunde). Sie fügen t-RNA an die m-RNA, verknüpfen die Aminosäuren
nebeneinander stehender t-RNA und lösen sie dann von der jeweils
vorangegangenen t-RNA ab.
Synthese von Ribosomen: Fünf
Chromosomenpaare des Zellkerns enthalten die zur Synthese ribosomaler
RNA nötigen Bauanleitungen (jedes dieser Chromosomen enthält zahlreiche
Duplikate der rRNA-Gene - die Zellen benötigen viel rRNA für ihre
Funktionen). Zur Transkription der rRNA-Gene versammeln sich diese
Chromosomen im Nukleolus (dessen Größe vom Ausmaß der rRNA-Synthese
abhängt). Es entstehen Untereinheiten der Ribosomen, die durch Poren
der Kernhülle transportiert und erst im Zytoplasma zu fertigen
Ribosomen zusammengestellt werden (Proteinsynthese erfolgt nur im
Zytoplasma, nicht im Zellkern).
Die Position eines Ribosoms in der Zelle hängt von der Natur des Proteins ab, das von ihm gerade übersetzt wird: Wird ein zytosolisches Protein translatiert, befindet sich das Ribosom im Zytosol; wird ein Membran- oder sekretorisches Protein synthetisiert, lagert es sich an die Membran des rauen endoplasmatischen Retikulums,
sodass das Proteinmolekül in die Zellmembran integriert bzw. nach
extrazellulär befördert werden kann.
Etwa 1% der RNA-Menge der Zelle sind sRNA (small RNAs) - kurze, meist nichtcodierende RNA, weniger als 200 Nukleotide lang, die im Zellkern (nukleäre sRNA) und Zytoplasma vorkommen. Man kennt zahlreiche sRNAs, darunter
 Mikro-RNA (miRNA, µRNA)
- aus 21-23 Nukleotiden bestehende RNA-Fragmente, welche die Expression
ihrer Zielgene stoppen können (Gen-Stilllegung durch RNA-Interferenz,
ein phylogenetisch sehr alter Mechanismus). Sie setzen sich auf
komplementäre DNS-Sequenzen einer mRNA und lassen sie auf verschiedene Art und Weise "verstummen" (gene silencing), indem sie z.B. die Translation behindern (beim Menschen ist die mRNA-Destabilisierung der primäre Wirkungsmechanismus):
Mikro-RNA (miRNA, µRNA)
- aus 21-23 Nukleotiden bestehende RNA-Fragmente, welche die Expression
ihrer Zielgene stoppen können (Gen-Stilllegung durch RNA-Interferenz,
ein phylogenetisch sehr alter Mechanismus). Sie setzen sich auf
komplementäre DNS-Sequenzen einer mRNA und lassen sie auf verschiedene Art und Weise "verstummen" (gene silencing), indem sie z.B. die Translation behindern (beim Menschen ist die mRNA-Destabilisierung der primäre Wirkungsmechanismus):
 Abbildung: Steuerung der Genexpression durch miRNA
Abbildung: Steuerung der Genexpression durch miRNA
Nach einer Vorlage bei Guyton and Hall, Textbook of Medical Physiology, 15th ed. Elsevier 2026
Primäre Mikro-RNA (pri-miRNA) ist das direkte Transkript des miRNA-Gens. Ein Mikroprozessor-Proteinkomplex (bestehend aus einer Ribonuclease und einem RNA-bindenden Protein) verwandelt es in prae-miRNA
- eine aus 70 Nukleotiden bestehende Schleifenstruktur. Diese gelangt
durch die Kernmembran in das Zytoplasma und wird durch die Endoribonuclease Dicer
(to dice = in Würfel schneiden) in kürzere doppelsträngige
DNA-Fragmente geschnitten. Dicer hilft auch, den aus mehreren Proteinen
(inklusive ein Riboprotein) bestehenden Molekülkomplex RISC (RNA-induced silencing complex)
zu aktivieren - der Komplex aus RISC und einem miRNA-Strang setzt sich
auf die mRNA (komplementäre RNA-Sequenz), hemmt ihre Translation und fördert ihren Abbau (RISC wirkt auch als Endonuclease) - die Konzentration des entsprechenden Proteins in der Zelle nimmt ab
 Man nimmt an, dass miRNAs auf die in der
Man nimmt an, dass miRNAs auf die in der  Abbildung
geschilderten Weise eine wichtige Rolle für die Steuerung des
Zellstoffwechsels spielen und Veränderungen ihrer Funktion diverse
Krankheitsbilder bedingen.
Abbildung
geschilderten Weise eine wichtige Rolle für die Steuerung des
Zellstoffwechsels spielen und Veränderungen ihrer Funktion diverse
Krankheitsbilder bedingen.
 Kleine Kern- (small nuclear) RNA (snRNA)
sind am Spleißen von prä-mRNA zu mRNA beteiligt. Sie bestehen aus 20-25
Nikleotiden, die ebenfalls mit der Genexpression interferieren. Soll die Expression eines Proteins (dessen genetische DNA-Basensequenz bekannt ist) beeinflusst werden, wäre dies mittels entsprechend komplementärer snRNA möglich.
Kleine Kern- (small nuclear) RNA (snRNA)
sind am Spleißen von prä-mRNA zu mRNA beteiligt. Sie bestehen aus 20-25
Nikleotiden, die ebenfalls mit der Genexpression interferieren. Soll die Expression eines Proteins (dessen genetische DNA-Basensequenz bekannt ist) beeinflusst werden, wäre dies mittels entsprechend komplementärer snRNA möglich.
 Kleine Interferenz-RNA (siRNA, small interfering RNA), auch silencing RNA, 20-25 Basenpaare lang, funktionieren ähnlich wie Mikro-RNA - sie
können die Translation von Zielgenen blockieren. Auch sie nutzen den
Dicer-Mechanismus, können aber auch die Genexpression fördern.
Kleine Interferenz-RNA (siRNA, small interfering RNA), auch silencing RNA, 20-25 Basenpaare lang, funktionieren ähnlich wie Mikro-RNA - sie
können die Translation von Zielgenen blockieren. Auch sie nutzen den
Dicer-Mechanismus, können aber auch die Genexpression fördern.
 Zahlreiche weitere sRNA sind bekannt, z.B. Piwi-interacting RNA (piRNA - Abkürzung für P-element induced wimpy testis) - beim Menschen notwendig für die Spermatogenese; kleine nukleoläre RNA (snoRNA, small nucleolar RNA) - machen Ribosomen fit für die Translation und snRNAs für das Splicing; etc.
Zahlreiche weitere sRNA sind bekannt, z.B. Piwi-interacting RNA (piRNA - Abkürzung für P-element induced wimpy testis) - beim Menschen notwendig für die Spermatogenese; kleine nukleoläre RNA (snoRNA, small nucleolar RNA) - machen Ribosomen fit für die Translation und snRNAs für das Splicing; etc.
Translation
Die m-RNA wird wie ein Datenstreifen durch die Ribosomen gezogen und die Information so umgesetzt. Translation ist die "Übersetzung" von der Nukleinsäure- in die
Aminosäure-Sprache.


Abbildung: Schema der Eiweißsynthese
Nach einer Vorlage in Panini SR, Medical Biochemistry, 2nd ed. 2021 (Thieme)
Am
Start-Codon fügen sich Ribosomen aus ihren Untereinheiten zusammen und
beginnen die Proteinsynthese, indem sie Aminosäure für Aminosäure zu
Peptidketten zusammensetzen.
A: Ein Signalpeptid
(rot dargestellt) am Beginn der entstehenden Peptidkette leitet einen
Vorgang ein, der zur Anlagerung des Ribosoms an das raue
endoplasmatische Retikulum führt (vgl. weiter unten). Ein Ribonukleoprotein namens signal-recognition particle (SRP, Signalerkennungspartikel) bindet an die Startsequenz (Signalsequenz) des entstehenden Proteins. Das stoppt die Proteinsynthese (translation arrest), bis der SRP-Signalsequenz-Komplex an einen freien SRP-Rezeptor (docking protein) in der Membran des endoplasmatischen Retikulums bindet.
B: Das SRP bindet an den SRP-Rezeptor und verankert dabei das Ribosom an einem Translocon
in der Membran des endoplasmatischen Retikulums. Dieses bietet einen
Durchlass für die wachsende Peptidkette in Richtung Lumen des
sarkoplasmatischen Retikulums.
C: Das neu zusammengesetzte Peptid nimmt an Länge zu...
D: ...und wird in das Lumen des endoplasmatischen Retikulums abgegeben.
E: Erreicht ein Stop-Codon das Ribosom, dissoziiert dieses und die Translation ist beendet. Luminale Enzyme trennen das Signalpeptid vom Proprotein ab. Das Proprotein wird anschließend via Modifikation im endoplasmatischen Retikulum oder im Golgi-Apparat zum endgültigen Peptid / Protein

Die Proteinsynthese eukaryoter Zellen beginnt immer mit Methionin (Starter-Aminosäure).
Mitochondrien (und Bakterien) benutzen hingegen Formylmethionin als
Starter der Translation (dabei entstehen formylierte Proteine).
Bei der Eiweißsynthese - also der korrekten Aneinanderreihung von Aminosäuren entsprechend dem genetischen Bauplan - gilt der "genetische Code": Bestimmte Basen-Dreiergruppen (Tripletts) veranlassen
das Arrangement entsprechender Aminosäuren.
Translation erfolgt im Zellplasma (für zytoplasmatische
Proteine) bzw. in der Wand des endoplasmatischen Retikulums (für
Membran- und Sekretproteine) - s. <Animation. Man schätzt, dass pro
Sekunde 6-8 Aminosäuren an die entstehende Aminosäurenkette angefügt
werden. Dieser rasche Vorgang ist nicht frei von Defekten, und etwa
jedes fünfte Proteinmolekül wird fehlerhaft gefaltet. Solche
"verkrüppelten" Eiweißmoleküle werden vorzugsweise von Proteasomen wieder abgebaut.
Steuerung der Produktion: Die Produktionsrate von Proteinen ist reguliert - durch (In-) Aktivierung von Translations-Initiationsfaktoren. Nach Fertigstellung der Proteinmoleküle können diese weiter (posttranslational) modifiziert werden.
Protein sorting
Wohin gelangen die neu gebildeten Proteine? Das ist eine entscheidende Frage, denn der gerichtete Transport (protein sorting) an bestimmte "Adressen" in der Zelle erfordert unterschiedliche Mechanismen. Die
Biosynthese von Proteinen beginnt grundsätzlich an Ribosomen im
Zytoplasma; sie kann hier komplett ablaufen oder in der Wand des rauen
endoplasmatischen Retikulums abgewickelt werden:
 Der zytoplasmatische Weg (cytoplasmic pathway) dient
der Synthese von Eiweiß für Zellkern, Zytoplasma, Mitochondrien und
Peroxisomen. Spezifische Translokationssignale fördern die Proteinbildung für Zellkern, Mitochondrien oder Peroxisomen - oder ein
Translokationssignal fehlt überhaupt, und das Protein wird direkt in das Zytoplasma
abgegeben, wo die gesamte Translation (Proteinsynthese) abläuft.
Der zytoplasmatische Weg (cytoplasmic pathway) dient
der Synthese von Eiweiß für Zellkern, Zytoplasma, Mitochondrien und
Peroxisomen. Spezifische Translokationssignale fördern die Proteinbildung für Zellkern, Mitochondrien oder Peroxisomen - oder ein
Translokationssignal fehlt überhaupt, und das Protein wird direkt in das Zytoplasma
abgegeben, wo die gesamte Translation (Proteinsynthese) abläuft.
 Der sekretorische Weg (secretory pathway)
dient der Synthese von Sekreten oder von integralen Membranproteinen, Eiweiß für das endoplasmatische Retikulum oder für
Lysosomen. Für diesen Weg
rücken Ribosomen an die Wand des rauen endoplasmatischen Retikulums:
Der sekretorische Weg (secretory pathway)
dient der Synthese von Sekreten oder von integralen Membranproteinen, Eiweiß für das endoplasmatische Retikulum oder für
Lysosomen. Für diesen Weg
rücken Ribosomen an die Wand des rauen endoplasmatischen Retikulums:
Sekretorischer Weg - Transfer in raues EPR
Zur Sekretion bestimmte Proteine (Hormone, Transmitter, Mediatoren, Enzyme..) werden durch die Membran von Vesikeln in deren Inneres verbracht, integrale Membranproteine werden in der betreffenden Lipid-Doppellamelle verankert (solche Moleküle haben lipophile α-helikale Domänen, mit
denen sie sich in die Membran "verzapfen"). Proteine haben stark hydrophile (elektrisch geladene) Sequenzen und verhalten sich insgesamt lipophob. Ihr Transport in oder durch Lipidmembranen bedarf spezieller Abläufe im Rahmen des "sekretorischen Weges".
Proteine, die
über den sekretorischen Weg produziert werden, verfügen an ihrem
"Kopfende" über eine 15-60 Aminosäuren lange Sequenz, die als Signalpeptid
bezeichnet wird und die Anlagerung an das endoplasmatische Retikulum
vermittelt. Diese Sequenz hat nahe ihrem N-Terminus eine stark basische
Aminosäure und daran anschließend eine stark hydrophobe Folge von 10-15
Aminosäuren. Dieses Signalpeptid bindet an ein Signalerkennungspartikel (SRP, signal-recognition particle), ein
Ribonukleoprotein, das seinerseits an ein Ribosom andockt und den so
entstandenen Ribosom-mRNA-Peptidkomplex an die Membran des
endoplasmatischen Retikulums fixiert. Auf diese Weise kann die
wachsende Polypeptidkette (das Pro-Protein) bei der Translation in das Lumen des EPR (endoplasmatischen Retikulums) gelangen, wo das Signalpeptid schließlich enzymatisch abgespalten wird ( Animation).
Animation).

 Animation: Ablauf der Synthese eines für den Export bestimmten Proteins
Quelle: Wikipedia / Bensaccount
Animation: Ablauf der Synthese eines für den Export bestimmten Proteins
Quelle: Wikipedia / Bensaccount
U.a. ist dargestellt, wie Proteine in Membranen integriert werden. So
verfügen z.B. für das endoplasmatische Retikulum bestimmte Proteine
über eine spezifische Sequenz, die von einem Protein-RNA-Komplex (SRP: Signal Recognition Particle)
erkannt wird.
Der resultierende SRP-Peptid-Ribosom-Komplex wird dann am
endoplasmatischen Retikulum erkannt und gebunden, die Proteinbildung setzt
sich durch die Membran fort

Dieses "durch die Membran stecken" ist ein energetisches Problem, das
aufwändig gelöst wird. So befördert das endoplasmatische Retikulum für
die Sekretion bestimmte Aminosäureketten über einen in der  Abbildung dargestellten Mechanismus schrittweise in und durch die
Membran hindurch. Die Kanäle (Translocons),
durch welche die aus den Ribosomen wachsenden - hydrophilen -
Aminosäureketten "gesteckt" (transloziert) werden, ersparen diesen
einen direkten Kontakt mit hydrophobem Membranmaterial.
Abbildung dargestellten Mechanismus schrittweise in und durch die
Membran hindurch. Die Kanäle (Translocons),
durch welche die aus den Ribosomen wachsenden - hydrophilen -
Aminosäureketten "gesteckt" (transloziert) werden, ersparen diesen
einen direkten Kontakt mit hydrophobem Membranmaterial.
Die notwendige Verknüpfung der Expression einer Signalsequenz
einerseits mit der Verfügbarkeit eines SRP-Rezeptors andererseits - ist
kein SRP frei, stoppt die Proteinsynthese (translation arrest,  Abbildung
- stellt sicher, dass die Synthese von Membran- und sekretorische
Proteine nur dann fortgesetzt wird, wenn für sie ein
Translokationskanal (ein Translocon) frei ist (cotranslational translocation).
Abbildung
- stellt sicher, dass die Synthese von Membran- und sekretorische
Proteine nur dann fortgesetzt wird, wenn für sie ein
Translokationskanal (ein Translocon) frei ist (cotranslational translocation).

 Abbildung: Herstellung eines sekretorischen Proteins (vgl. <Animation)
Abbildung: Herstellung eines sekretorischen Proteins (vgl. <Animation)
Nach einer Vorlage bei Boron / Boulpaep, Medical Physiology, 1st ed., Saunders 2003
Ein Ribonukleoprotein namens signal-recognition particle (SRP) bindet an die Startsequenz des entstehenden Proteins (Signalsequenz). Das stoppt die Proteinsynthese (translation arrest, Schritt 2), bis der SRP-Signalsequenz- Komplex an einen freien SRP-Rezeptor (docking protein) in der Membran des endoplasmatischen Retikulums bindet.
Dann gelangt die entstehende Aminosäurekette durch dessen Membran (Translokation, einen betreffenden Kanal nennt man Translocon), und die Syntheseaktivität des Ribosoms wird fortgesetzt (Schritt 3).
Eine Signalpeptidase
spaltet die Signalsequenz ab (Schritt 4), diese verbleibt in der Membran des
endoplasmatischen Retikulums.
Die Eiweißsynthese schreitet fort, das Protein wird mit
Kohlenhydrat-Seitenketten komplettiert (Schritt 5); ist es fertiggestellt, löst es
sich vom Translokationsapparat und steht für Sekretionsvorgänge zur
Verfügung, das Ribosom zerfällt in seine zwei Bestandteile (Schritt 6)

Viele Proteine
werden nach ihrer Synthese modifiziert (sulfatiert, amidiert,
phosophatiert, gespalten etc), bevor sie in den Zellstoffwechsel
einmünden oder in den Extrazellulärraum exportiert werden
( Abbildung). Dann hat das Protein seine endgültige Form erhalten
und kann exozytiert werden (ein Vorgang, der durch Calciumionen
angeregt wird).
Abbildung). Dann hat das Protein seine endgültige Form erhalten
und kann exozytiert werden (ein Vorgang, der durch Calciumionen
angeregt wird).
 Um
Proteine in Vesikel zu bringen (von wo sie exozytiert werden können),
bedarf es der Beteiligung des rauen endoplasmatischen Retikulums
(Proteinsynthese) und des Golgi-Apparats (Verbringung in Vesikel).
Um
Proteine in Vesikel zu bringen (von wo sie exozytiert werden können),
bedarf es der Beteiligung des rauen endoplasmatischen Retikulums
(Proteinsynthese) und des Golgi-Apparats (Verbringung in Vesikel).
Die Konstruktion eines integralen Membranproteins
läuft etwas komplizierter ab - das Molekül löst sich nicht von der
Membran (wie sekretorische Proteine), sondern wird mit einem
hydrophoben Mittelteil in diese integriert. Dabei spielen spezielle
Aminosäuresequenzen ("Start-Transfer", "Stop-Transfer") eine steuernde Rolle.
Das Protein wird u.U. mehrfach durch die Membran gesteckt (abhängig
davon, wie viele transmembranale Sequenzen aufgebaut werden), ohne dass
es dabei die Membran verlässt.
Eiweißmoleküle, die in die Membran von Mitochondrien oder Peroxisomen
eingebaut werden, entstehen durch peroxisomale bzw. mitochondriale Synthesemechanismen.

 Abbildung: Synthese, Sortierung und Freisetzung von Peptiden
Abbildung: Synthese, Sortierung und Freisetzung von Peptiden
Nach einer Vorlage in Ritter / Flower / Henderson / Loke / MacEwan / Rang, Rang & Dale's Pharmacology, 9th ed. Elsevier 2020
Ribosomen
fädeln synthetisierte Peptide in das raue endoplasmatische Retikulum
ein, wobei bestimmte dreidimensionale Strukturen entstehen. Dann
gelangen diese in das glatte EPR und werden via Transportvesikel (mit
gemischtem Inhalt) in den Golgi-Apparat integriert.
Der Golgi-Apparat
nimmt eine Sortierung vor und reicht Vesikel - separiert mit jeweils
definiertem Inhalt - zur Exozytose weiter. Im Zuge dieses vesikulären
Transports können molekulare Modifikationen vorgenommen werden.
Die Sekretion
kann in gesteuerter Weise erfolgen (z.B. die Freisetzung von Zytokinen
durch Immunzellen oder die eines Transmitters an einer Synapse) oder
läuft konstitutiv ab (z.B. Freisetzung von Plasmaproteinen durch
Leberzellen). Konstitutive Synthese und Sekretion von Peptiden /
Proteinen erfolgt kontinuierlich und bedarf keiner wesentlichen
Zwischenspeicherung; regulierte Sekretion hingegen greift auf in
Vesikeln gespeicherte Proteine zurück

Damit die anschließende
räumliche Faltung zu funktionsfähigen Proteinmolekülen richtig erfolgt,
treten weiters Chaperone auf den Plan. Chaperone stellen sicher, dass neu synthetisierte Proteine ihre funktionelle Struktur erhalten. Weiters kümmern sie sich darum, dass Proteinmoleküle,
die - unvernetzt, aber funktionslos - durch Tunnelmoleküle in Membranen (z.B. von
Mitochondrien) treten, nachher wieder zurückgefaltet werden und ihre
Funktion wiedererlangen.
 Wie
können sich Zellen auf Veränderungen der Umwelt anpassen? Ein Mechanismus besteht darin, die Transkription und Translation
zu steuern und/oder deren molekularen Ergebnisse (Ribonukleinsäure,
Protein) zu modifizieren. Nur ein kleiner Teil der vorhandenen Gene
wird auch exprimiert. Die Exprimierungsmuster können wechseln (s. oben unter "Genexpression").
Wie
können sich Zellen auf Veränderungen der Umwelt anpassen? Ein Mechanismus besteht darin, die Transkription und Translation
zu steuern und/oder deren molekularen Ergebnisse (Ribonukleinsäure,
Protein) zu modifizieren. Nur ein kleiner Teil der vorhandenen Gene
wird auch exprimiert. Die Exprimierungsmuster können wechseln (s. oben unter "Genexpression").
Die Steuerung von Transkriptionsvorgängen im Zellkern durch von außen
auf die Zelle einwirkende Einflüsse erfolgt im Rahmen von
Signalübermittlungen, wie sie z.B. nach Bindung von Hormonen an ihre
Rezeptoren auftreten - etwa durch second messengers, den JAK-STAT- oder einen Ras-abhängigen Weg.
Ontogenese und Zellspezifität: Die einzelnen Zelltypen des Organismus regulieren die Transkriptionsvorgänge unterschiedlich
- sie exprimieren bestimmte Sets von Transkriptions-Regulatoren.
Während der Entwicklung schalten solche Sets spezifische
Regulator-Kombinationen ein und aus. Diese Muster bewirken die
Unterschiedlichkeit der Zelltypen im reifen Organismus.
Gentechnik nützt Mechanismen der Vorgänge bei Entstehung und
Wirkung von Nukleinsäuren ("Genbanken"). Da alle Lebewesen
gleichen genetischen Prinzipien folgen, kann man z.B. Bakterien- oder
Hefezellen menschliche Gene in ihr Erbgut einschleusen und sie zur
Bildung großer Mengen identischen Proteins (Hormone,
Gerinnungsfaktoren, Immunstoffe, Wachstumsfaktoren etc.) veranlassen.
Wachstumsfaktoren (Growth factors, GF) sind Glykoproteine (auch einige Steroide) mit hoher Wirksamkeit (nano- bis pikomolarer Bereich, d.h. zwischen 10-12 und 10-9 M/l). Es sind Zytokine, die spezifisch das Wachstum und die Differenzierung bestimmter Zielzellen steuern. Sie können von vielen verschiedenen Zellarten produziert werden; einige
können auch Zellteilungen auslösen, sind also Mitogene. Ihr Blutspiegel
ist unter Basisbedingungen sehr niedrig, kann aber bei entsprechender
Stimulation innerhalb weniger Stunden bis zu tausendfach ansteigen. Zu
den Wachstumsfaktoren zählt man auch hämatopoetisch wirkende Proteine (colony stimulating factors,  s. dort).
s. dort).
In einem vielzelligen Organismus sind Wachstum und Teilung der Zellen
im Allgemeinen restringiert, d.h. ohne die Anwesenheit extrazellulärer
Signale (Wachstumsfaktoren) ist die Zelle in einem Gleichgewichtszustand "eingefroren". Inkubiert man Zellen in vitro mit Wachstumsfaktoren, beginnen
diese nach einigen Stunden mit der Synthese von DNA (Synthesephase).
In der Regel reagieren Zellen nicht auf die Anwesenheit nur eines Wachstumsfaktors, sondern auf
bestimmte Kombinationen davon - es bedarf also eines
Wachstumsfaktor-Musters, um Zellen zur Mitose zu veranlassen.
Umgekehrt gehen somatische Zellen in die G0-Phase über, wenn ihnen Wachstumsfaktoren entzogen werden - und nach einiger Zeit ohne Wachstumsfaktoren kann es zu Apoptose kommen.
Wachstumsfaktoren wirken über Rezeptoren, die meist Tyrosinkinaseaktivität haben. Auf diesem Wege bewirken sie die Inaktivierung (Phosphorylierung) von Rb-Protein, was die Freisetzung des Transkriptionsfaktors E2-F zur Folge hat (Kontrollfaktoren, Einleitung der S-Phase).
 Als Rb-Protein (pRB,
Retinoblastom-Protein) bezeichnet man ein physiologisches Protein, das
in den Zellzyklus regulierend eingreift, indem es die Aktivität von Transkriptionsfaktoren unterdrückt: Es verhindert u.a. übertriebenes Zellwachstum, indem es den Teilungszyklus
aufhält, bis die Zelle bereit für eine Mitose ist. Auf diese Weise kann
es auch die Bildung von Tumoren verhindern (der Name stammt daher, dass
Mutationen des Rb-Proteins zur Bildung von Retinoblastomen führen
kann).
Als Rb-Protein (pRB,
Retinoblastom-Protein) bezeichnet man ein physiologisches Protein, das
in den Zellzyklus regulierend eingreift, indem es die Aktivität von Transkriptionsfaktoren unterdrückt: Es verhindert u.a. übertriebenes Zellwachstum, indem es den Teilungszyklus
aufhält, bis die Zelle bereit für eine Mitose ist. Auf diese Weise kann
es auch die Bildung von Tumoren verhindern (der Name stammt daher, dass
Mutationen des Rb-Proteins zur Bildung von Retinoblastomen führen
kann).
Der Wachstumsfaktor BDNF (Brain-derived neurotrophic factor) schützt Nervenzellen und Synapsen und fördert deren Wachstum. Er ist ein mit Nervenwachstumsfaktoren eng verwandtes Neurotrophin
(hierher gehören auch der Nerven-Wachstumsfaktor NGF). Großhirnrinde
und Hippokampus sind besonders reich an BDNF-Rezeptoren. Chronischer Stresseinfluss reduziert die Bildung von BDNF und senkt dadurch seine neurotrophe und Reparaturaktivität.
BDNF spielt beim Aufbau von Gedächtnisspuren
eine Rolle - der angeregte postsynaptische Apparat setzt es frei, es
diffundiert zum präsynaptischen Teil, bindet an Rezeptoren und
moduliert die Transmitterfreisetzung, z.B. durch Einfluss auf den Calciumeinstrom: Nimmt dieser zu, werden Vesikel mobilisiert, die
Transmitterfreisetzung vermehrt und die synaptische Effizienz
gesteigert.
Mutationen des
BDNF-Gens bewirken das Undine-Syndrom (kongenitales zentrales
Hypoventilationssyndrom), das durch gestörte CO2-Chemorezeptorsensitivität gekennzeichnet ist.
BDNF wird nicht nur im Gehirn, sondern auch in der Peripherie gebildet (u.a. von aktiven Muskelzellen,
es regt den Muskelaufbau an und umgekehrt wird seine Bildung durch
Muskelaktivität stimuliert - sein Blutspiegel steigt dabei rasch an und
nimmt nach der Muskeltätigkeit ebenso zügig wieder zum Ruhewert ab).

 Abbildung: EGF-Rezeptor-Wirkungen an Zielorganen
Abbildung: EGF-Rezeptor-Wirkungen an Zielorganen
Modifiziert
nach Chen J, Zeng F, Forrester SJ, Eguchi S, Zhang MZ, Harris RC.
Expression and Function of the Epidermal Growth Factor Receptor in
Physiology and Disease. Physiol Rev 2016; 96: 1025-69
Der
epidermale Wachstumsfaktor wirkt wachstumsfördernd, wundheilend und
differenzierend an Nervensystem, Herz und Lunge, Nieren, Haut
(inklusive Brustdrüse) und Knochen, sowie an der Leber

Der epidermale Wachstumsfaktor (Epidermal growth factor, EGF) wirkt - wie auch der Transforming Growth Factor TGF-α - auf Zellen, die EGF-Rezeptoren
( Abbildung) exprimieren (davon gibt es zahlreiche Spielarten), wachstumsfördernd
und mitosetriggernd (EGF wird für
Zellkulturen verwendet). EGF kommt in zahlreichen Gewebsflüssigkeiten
vor, z.B. auch im Speichelsekret, und hat dort trophische Wirkungen.
EGF spielt für die Wundheilung eine wichtige Rolle: so wird er in
heilenden Hautwunden von Keratinozyten, Makrophagen und anderen
eingewanderten Entzündungszellen produziert.
Abbildung) exprimieren (davon gibt es zahlreiche Spielarten), wachstumsfördernd
und mitosetriggernd (EGF wird für
Zellkulturen verwendet). EGF kommt in zahlreichen Gewebsflüssigkeiten
vor, z.B. auch im Speichelsekret, und hat dort trophische Wirkungen.
EGF spielt für die Wundheilung eine wichtige Rolle: so wird er in
heilenden Hautwunden von Keratinozyten, Makrophagen und anderen
eingewanderten Entzündungszellen produziert.
Zur Gruppe der EGF zählt auch der Heparin-binding EGF-like growth factor
(HB-EGF); er moduliert mehrere Zellaktivitäten und findet sich
besonders im ZNS (Neuronen und Gliazellen). Nach Ischämie /
Sauerstoffmangel im Gehirn wird er stark exprimiert und regt die
Neurogenese an.
Zu den Transformierenden Wachstumsfaktoren (Transforming growth factors TGF)
werden zwei Peptidklassen gezählt: TGF-α und TGF-β; diese sind
unterschiedlich aufgebaut und wirken über verschiedene Rezeptoren.
TGf-α wird von Makrophagen, Gehirn- und Hautzellen gebildet und fördert das Epithelwachstum.
TGF-β
(3 Subtypen: 1-3) regulieren den Zellzyklus und induzieren Apoptose,
beeinflussen Zelldifferenzierung, Entwicklung, Wachstum und
Regeneration, sowie das Immunsystem. TGF-β
hemmt das Wachstum der meisten Epithelzellen, regt die Gewebsbildung
(Kollagensynthese, Proteoglykanbildung u.a.) an und wirkt
entzündungshemmend, gleichzeitig regt es bestimmte Immunfunktionen an,
wie die IgA-Produktion in der Darmschleimhaut.
Es gibt eine Superfamilie von strukturell ähnlich aufgebauten Molekülen, die als TGF-β Superfamilie (Transforming growth factor-ß superfamily) zusammengefasst werden. Dazu gehören außer TGF-ß1 bis TGF-ß5 u.a. BMPs (knochenmorphogenetische Proteine), Inhibin, und das "Anti-Müller-Hormon" MIF.
Gefäßwachstumsfaktoren (Vascular endothelial growth factor VEGF
- A bis F) entstehen in zahlreichen Geweben, in höherer Menge vor allem in Nierenglomerula (Podozyten) und Herzmuskelzellen.
Sauerstoffmangel (dieser induziert den Transkriptionfaktor HIF
- Hypoxie-induzierter Faktor -, welcher die Synthese von VEGF
reguliert) sowie andere Faktoren (TGF, PDGF) regen die Produktion von
VEGF an. Sie wirken an verschiedenen Orten, vor allem den Gefäßen, über
VEGF-Rezeptoren (VEGFR 1 bis
3), die Tyrosinkinasen sind und Kaskaden von Proteinkinasen aktivieren;
diese aktivieren wiederum Transkriptionsfaktoren, die bei der
Angiogenese benötigte Gene ablesen.
VEFG führen zu
Gefäßneubildung (Angiogenese)
inklusive Lymphangiogenese,
fördern die Bildung von
Stickstoffmonoxid und damit Vasodilatation und Blutdrucksenkung,
erhöhen die Gefäßpermeabilität, beteiligen sich an
Wundheilungsprozessen. VEGF-A bewirkt das Aussprossen neuer
Blutkapillaren, VEGF-C lymphatischer Gefäße.
Der Nervenwachstumsfaktor (Nerve growth factor NGF) wirkt neurotroph
- insbesondere in der Embryonalzeit, wo er vorwachsenden
Axonen den Weg zu "richtigen" Dendriten und synaptischen Verschaltungen
weist. NGF lässt Nervenzellen wachsen, aber nicht teilen. NGF gehört in die Gruppe der Neurotrophine
- basischer Proteine (~13 kDa), welche Verbindungen zwischen
Nervenzellen herstellen und ihren Fortbestand sichern. In diese Gruppe
gehört auch der brain-derived neurotrophic factor BDNF:
Dieser ist vor allem im limbischen System und Großhirn dort aktiv, wo
Gedächtnis und abstraktes Denken entstehen (Langzeitgedächtnis!).
 Für die Entdeckung des NGF erhielten die italienische Neurobiologin Rita Levi-Montalcini und der US-Amerikaner Stanley Cohen 1986 den Nobelpreis für Physiologie oder Medizin.
Für die Entdeckung des NGF erhielten die italienische Neurobiologin Rita Levi-Montalcini und der US-Amerikaner Stanley Cohen 1986 den Nobelpreis für Physiologie oder Medizin.
Blutplättchen-Wachstumsfaktoren (Platelet derived growth factors PDGF)
werden von Thrombozyten (Speicherung in, und Freisetzung aus Granula),
aber auch von Makrophagen, Endothelzellen und glatten Muskelzellen
gebildet (PDGF-A bis PDGF-D). Beispielsweise setzen Thrombozyten bei Verletzung PDGF-A frei, dieses regt das Zellwachstum in umliegendem Gewebe an.

 Abbildung: Wirkungen des PDGF
Nach Donovan J, Abraham D, Norman J. Platelet-derived growth factor signaling in mesenchymal cells. Front Biosci 2013; 18: 106-19
Abbildung: Wirkungen des PDGF
Nach Donovan J, Abraham D, Norman J. Platelet-derived growth factor signaling in mesenchymal cells. Front Biosci 2013; 18: 106-19
Die
Wirkungen umfassen Migration, Proliferation, Differenzierung, Überleben
und Bildung von extrazellulärem Matrixmaterial.
Zielzellen sind u.a.
Fibroblasten, Gefäßmuskelzellen, Osteoblasten, Osteoklasten,
Chondrozyten
 PDGFs wirken auf verschiedenste Zellen, auch im Rahmen
der Embryogenese (ZNS, Blutgefäße, Nieren,
Lungen), sowie auf Zellproliferation, Zellmigration (Monozyten,
Fibroblasten u.a.), Wundheilung und Angiogenese (
PDGFs wirken auf verschiedenste Zellen, auch im Rahmen
der Embryogenese (ZNS, Blutgefäße, Nieren,
Lungen), sowie auf Zellproliferation, Zellmigration (Monozyten,
Fibroblasten u.a.), Wundheilung und Angiogenese ( Abbildung).
Abbildung).
Fibroblasten-Wachstumsfaktoren (Fibroblast growth factor FGF - eine Proteinfamilie mit 23 bekannten Mitgliedern, FGF-1 bis FGF-23)
spielen wichtige Rollen für Wachstum (Proliferation) und
Differenzierung an
zahlreichen Geweben. Sie wirken auf Angiogenese (Gefäßaussprossung),
Wundheilung (Epithelialisierung, Hämatopoese), embryonale Entwicklung
(Entwicklung von Muskel-, Lungen-, Leberzellen) und diverse endokrine
Signalwege.
Beim Menschen sind mehr als 20 FGF's bekannt. FGFs
sind mit der extrazellulären Matrix eng assoziiert und müssen an Heparansulfat-Proteoglykane interagieren,
um Rezeptoren in der Membran von Zielzellen aktivieren zu können.
Transmembran- und intrazelluläre Proteine beteiligen sich an der
Regulation der Effekte von FGFs in Zielzellen.
FGF19 ist an Morphogenese, Wachstum und Entwicklung des Embryos sowie an Gewebereparatur beteiligt.
Bei Einwirkung von Gallensäuren auf spezielle (Farnesoid-) Rezeptoren
(FXR) wird FGF19 von Zellen im Ileum (distaler Dünndarm) freigesetzt
und entspannt nach Passage des Pfortaderkreislaufs die Wand der Gallenblase, sodass diese sich leichter füllen kann.
FGF23 wird von Osteoblasten gebildet und beteiligt sich an der Steuerung des Calc
ium- und Phosphat-Stoffwechsels. Über
Wirkungen auf die DNA-Ablesung stimulieren FGFs die Zellproliferation,
sichern das Überleben der Zelle und fördern ihre Motilität.
Der Hepatozyten-Wachstumsfaktor (Hepatocyte growth factor / Scatter factor HGF/SF) ist ein Wachstums-, morphogener und Motilitätsfaktor, der über seinen Rezeptor c-MET
- auch über größere Distanz (endokrin) - wirkt. Er wird von
mesenchymalen Zellen (nicht-epitheliale Leberzellen, Endothel) und
Fibroblasten gebildet und wirkt parakrin-multifunktional auf
epitheliale (z.B. Hepatozyten, Gallengangsepithel, Lunge, Niere, Haut,
Brustdrüse), endotheliale und hämatopoetische Zellen.
HGF/SF ist an Zellregeneration, Wundheilung und Embryonalentwicklung
beteiligt (Leber, Niere, Gehirn, Muskeln: c-MET-defiziente
Knockout-Mäuse überleben nicht). Werden Teile der Leber
entfernt, dann steigt der
HGF-Spiegel im Blut bis mehr als 20-fach an - dies bewirkt
kompensatorisches Wachstum von Lebergewebe.
Insulinähnliche Wachstumsfaktoren (insulin-like growth factors, IGFs)
sind in ihrer Aminosäuresequenz dem Insulin ähnlich und werden u.a. in
Leberzellen gebildet. Sie bilden ein komplexes interzelluläres
Kommunikationssystem (IGF-Achse), das zwei Membranrezeptoren (IGF1R und
IGF2R), zwei Liganden, sechs IGF-Bindungsproteinen (IGFBP 1-6) und
einigen IGFBP-assoziierten Proteasen besteht.
Das aus 70 Aminosäuren aufgebaute IGF-1 heißt auch
Somatomedin C (SM-C), Hepatozyten bilden es auf Anregung durch hGH (Wachstumshormon). Es spielt vor allem während des Körperwachstums eine wichtige proliferierende Rolle.
 Näheres zu IGFs s. dort
Näheres zu IGFs s. dort
Wundheilung
 s. auch dort
s. auch dort
Eine Verletzung (Trauma) aktiviert das System der Blutstillung sowie das Kininsystem (Schmerz). Im Wundbereich wirken das angeborene sowie adaptive Immunsystem, bis eingedrungene Pathogene abgewehrt sind. Parallel dazu erfolgen Wundverschluss (hier spielen Makrophagen eine tragende Rolle), Gewebereparatur und Wundheilung. Zahlreiche Faktoren sind beteiligt, z.B. regt IL-13 Fibroblasten und Kollagensynthese an.

 Abbildung: Mechanismen, über welche die extrazelluläre Matrix und
Wachstumsfaktoren zusammenwirken, um Signalkaskaden in der Zelle zu
aktivieren
Abbildung: Mechanismen, über welche die extrazelluläre Matrix und
Wachstumsfaktoren zusammenwirken, um Signalkaskaden in der Zelle zu
aktivieren
Nach einer
Vorlage in Kumar / Abbas / Fausto / Aster, Robbin and Cotran's
Pathological Basis of Disease, 8th ed. Saunders / Elsevier 2010
Integrine binden extrazelluläre Komponenten und interagieren mit dem Zytoskelett an fokalen Adhäsionskomplexen, Proteinaggregaten inklusive Aktin, Vinculin, Paxillin und Talin (Proteinen,
welche die Zelle für den Aufbau von Zellkontakten benörigt).
Folge sind nukleäre Signale (Mitte) oder die Produktion von second messengers.
Die Anlagerung von Wachstumsfaktoren
an ihre Rezeptoren kann ebenfalls zytoplasmatische Mechanismen
aktivieren, die mit diesen Signalwegen überlappen. Integrine und
Wachstumsfaktor-Rezeptoren interagieren bei der Übertragung
extrazellulärer Signale auf Zellantworten (crosstalk).
Die Zelle integriert diese Einflüsse und münzt sie in Antworten wie
Proliferation, Differenzierung, Migration oder auch Apoptose um. Diese
Mechanismen ermöglichen die Aktivierung von Zellen, wie sie bei der
Wundheilung benötigt wird

Viele dieser Faktoren sind bei der Wundheilung
essentiell: Sie regen Überleben, Bewegung und Kontraktilität sowie
Differenzierung beteiligter Zellen an und fördern die Gefäßneubildung.
Die Wachstumsfaktoren aktivieren dabei die Transkription von Genen, die
in ruhenden Zellen nicht benötigt werden, u.a. solche, welche den
Eintritt in den Teilungszyklus sowie dessen Fortschritt kontrollieren.
Extrazelluläre Matrix -
Basalmembranen und Interstitium, aufgebaut aus Kollagen, Elastin,
Fibronektin, Laminin, Proteoglykanen und Hyaluronsäure u.a. mit eingelagerten
Zelladhäsionsproteinen und anderen regulatorischen Substanzen - spielt
für die Wundheilung eine eminente Rolle. Sie gibt Leitstrukturen und
mechanische Stützung ab, speichert regulatorische
Moleküle und Wachstumsfaktoren, und setzt diese bei Verletzungen frei.
Dadurch werden zelluläres Wachstum, Proliferation, Bewegung und
Differenzierung beeinflusst und beschleunigt.
Bei Heilungsvorgängen
wirken extrazelluläre Matrixstrukturen ordnend auf das Gewebe ein,
ohne diese Information sind die Zellen nicht in der Lage, komplexe
Organmorphologie zu rekonstruieren. Stattdessen bildet sich
Narbengewebe aus.

Oben:
Normalerweise blockiert die intakte Haut Wasserverlust nach außen und
das Eindringen von Mikroben, Chemikalien etc. in den Körper. Dendritische Zellen erkennen, verarbeiten und präsentieren Antigene an T-Lymphozyten.
Darunter: Bei Verletzung der
Epidermis erhöhen Keratinozyten ihre Proliferationsrate sowie
Zytokinproduktion und wandern seitwärts, um die Wundstelle mit frischem
Epithel zu bedecken. T-Zellen werden an die Zone der Verletzung
rekrutiert. Bleibt die Reparatur bzw. Barrierefunktion unvollständig,
stellt sich eine Entzündung ein

Die Heilung von Hautwunden kann in folgende drei, sich überlappende Phasen eingeteilt werden:
 Entzündung (inflammation)
Entzündung (inflammation) - ausgelöst durch Vorgänge der
Thrombozytenaktivierung und
Gerinnselbildung
 Proliferation
Proliferation

- das Gerinnsel dient als Matritze für mobile Zellen, die durch
freigesetzte
Zytokine und
Wachstumsfaktoren angelockt
werden; Fibroblasten wandern ein, Granulationsgewebe

formiert sich, das Fibringerinnsel wird durch
Fibrinolyse weggeräumt; Neutrophile
und
Makrophagen bauen nutzlos gewordene Stoffe ab, entfernen sie,
bekämpfen Bakterien und beteiligen sich an Aufbauprozessen; und es
kommt zu Re-Epithelialisierung
 Reifung (maturation)
Reifung (maturation) mit Neubildung extrazellulärer Matrix sowie Wundkontraktion
Bei ausgedehnten Wunden ist die
Entzündungsreaktion stärker ausgeprägt, das für 3-7 Tage zunehmende
Granulationsgewebe (einwachsende Fibroblasten und Gefäße mit hoher
Permeabilität) ausgebreiteter und ödematös, und es wird Kollagen
abgelagert, das dann zu einer Narbe wird.
Belastbarkeit verheilter Wunden.
Nach Entfernen der Nähte aus einer
Operationsnarbe eine Woche nach dem Eingriff entspricht die mechanische
Belastbarkeit der Wunde nur etwa 10% derjeniger unverletzter Haut.
Anschließend nimmt sie kontinuierlich zu. Dieser Zeitverlauf
spiegelt die Qualität molekularer Rekonstruktionsvorgänge im Gewebe
wider. Nach 1-3 Monaten erreicht die Belastbarkeit ~70-80% der
Maximalwerte gesunder Haut - die Organisation der Matrixstrukturen
(Kollagen etc) führt
nicht immer zu vollständiger restitutio ad integrum.
Zu den Faktoren, die den Verlauf und das Ausmaß der Wiederherstellung der Gewebeintegrität beeinflussen, gehören
 systemische
systemische:
Ernährung (z.B. Eiweiß- oder Vitaminmangel!), Stoffwechselzustand (z.B.
Diabetes mellitus!), Kreislauf (z.B. Arteriosklerose!), Hormonstatus
(z.B. Glukokortikoide!)
 lokale
lokale: Mechanische (Druck / Überdehnung an Wunde), Lage und Größe der Wunde, Fremdkörper, Infektionen
Wie weist man spezifisch Gene, Proteine, RNA nach?
 Western blots identifizieren Proteine: Diese Methode ermöglicht die Auftrennung von Zielproteinen nach ihrem Molekulargewicht. Antikörper
(monoklonal oder polyklonal) dienen als Sonden zur spezifischen
Bindung der Zielproteine.
Western blots identifizieren Proteine: Diese Methode ermöglicht die Auftrennung von Zielproteinen nach ihrem Molekulargewicht. Antikörper
(monoklonal oder polyklonal) dienen als Sonden zur spezifischen
Bindung der Zielproteine.

 Abbildung: Darstellung von Proteinen mittels Western-Blot
Nach einer Vorlage in Abbas / Lichtman / Pillai: Cellular and Molecular Immunology, 9th ed. 2018
Abbildung: Darstellung von Proteinen mittels Western-Blot
Nach einer Vorlage in Abbas / Lichtman / Pillai: Cellular and Molecular Immunology, 9th ed. 2018
Ein Proteingemisch (Zellhomogenat) wird mittels SDS (Sodium dodecyl sulfate) in einem Gel mittels Polyacrylamidgel- Elektrophorese (PAGE) aufgetrennt (SDS-PAGE, oben), die getrennten Fraktionen mittels nochmaliger Elektrophorese auf
eine Mikrozellulose-Membran übertragen (Mitte; die SDS wird dabei vom
Protein entfernt) und mit einem Antikörpercocktail behandelt (unten
links). Mit einem Fluorophor oder Enzym markierte Sekundärantikörper ermöglichen die Darstellung der markierten Fraktionen auf einer Fotoplatte (unten rechts)
Zellhomogenate werden durch
Gelelektrophorese mit einem anionischen Tensid - Natriumlaurylsulfat
(SDS) - denaturiert, linearisiert und negativ aufladen. Die
Wanderungsgeschwindigkeit der solchermaßen veränderten Proteine ist nun
umgekehrt proportional zu ihrem Molekulargewicht. Die entstandenen
"Blots" werden auf Mikrozellulose
übertragen, dabei von SDS befreit und auf dem Papier fixiert. Spezifische Primärantikörper werden aufgebracht, die an Epitope binden und anschließend mittels fluorophor- (genauer) oder enzymdotierter (Chemoluminiszenz) Sekundärantikörper detektiert
werden. Die Intensität des optischen Signals wird auf Photofilm festgehalten.
Sowohl die Konzentration als auch die Molekülgröße der Antigene wird
durch diese Methode ermittelt ( Abbildung).
Abbildung).
 Mittels (DNA-) Polymerase-Kettenreaktion (PCR: polymerase chain reaction) lassen sich DNA-Abschnitte in vitro vervielfältigen und dadurch molekulare Sonden herstellen. Mit deren Hilfe lassen sich DNA-Sequenzen präzise orten (DNA sequencing).
Mittels (DNA-) Polymerase-Kettenreaktion (PCR: polymerase chain reaction) lassen sich DNA-Abschnitte in vitro vervielfältigen und dadurch molekulare Sonden herstellen. Mit deren Hilfe lassen sich DNA-Sequenzen präzise orten (DNA sequencing).
 Die PCR ermöglicht eine rasche Kopie und Multiplikation spezifischer DNA-Sequenzen bis zu einer Länge von 1 kb (103 Basen) zu analytischen und präparativen Zwecken. Die Methode nutzt kurze, Endstücken komplementäre Oligonukleotid-Primer.
Die PCR ermöglicht eine rasche Kopie und Multiplikation spezifischer DNA-Sequenzen bis zu einer Länge von 1 kb (103 Basen) zu analytischen und präparativen Zwecken. Die Methode nutzt kurze, Endstücken komplementäre Oligonukleotid-Primer.
 Southern blots identifizieren Gene: Es werden Nukleinsäuresequenzen (Gensonden, hybridization probes)
hergestellt (PCR), die komplementär zu der gesuchten DNA-Sequenz sind.
Mit ihrer Hilfe können Gensequenzen z.B. in Zellhomogenaten aufgefunden
werden (Einzelheiten wie Gelelektrophorese, Blotting von Gel auf
Nitrozellulose, Hybridisierung und Sichtbarmachung s. Biochemie).
Southern blots identifizieren Gene: Es werden Nukleinsäuresequenzen (Gensonden, hybridization probes)
hergestellt (PCR), die komplementär zu der gesuchten DNA-Sequenz sind.
Mit ihrer Hilfe können Gensequenzen z.B. in Zellhomogenaten aufgefunden
werden (Einzelheiten wie Gelelektrophorese, Blotting von Gel auf
Nitrozellulose, Hybridisierung und Sichtbarmachung s. Biochemie).
 Northern blots identifizieren mRNA-Transkripte:
Diese Methode erlaubt es festzustellen, welche Gewebe ein bestimmtes
RNA-Transkript herstellen. Das Verfahren ist ähnlich wie beim
Southern-Blot (Homogenisierung fraglicher Zellen etc), nur werden RNA-
und nicht DNA-Abschnitte nachgewiesen.
Northern blots identifizieren mRNA-Transkripte:
Diese Methode erlaubt es festzustellen, welche Gewebe ein bestimmtes
RNA-Transkript herstellen. Das Verfahren ist ähnlich wie beim
Southern-Blot (Homogenisierung fraglicher Zellen etc), nur werden RNA-
und nicht DNA-Abschnitte nachgewiesen.
Will man wissen, wie die Expression der Ziel-RNA in Gewebeschnitten
lokalisiert ist (welche Zellen erzeugen die RNA?), wendet man in-situ-Hybridisierung an, bei der die Bindung der Gensonde in Gewebeschnitten erfolgt (und sichtbar gemacht wird).
 Immunozytochemie: Wie beim Nachweis von RNA, kann auch der Proteinnachweis - statt in Zellhomogenaten - in Gewebeschnitten
erfolgen und so die Lokalisierung der gesuchten Proteine im
untersuchten Gewebe (auch innerhalb von Zellen) sichtbar gemacht werden
- nur eben nicht mittels Hybridisierung, sondern mit Hilfe von
Antigen-Antikörper-Reaktionen.
Immunozytochemie: Wie beim Nachweis von RNA, kann auch der Proteinnachweis - statt in Zellhomogenaten - in Gewebeschnitten
erfolgen und so die Lokalisierung der gesuchten Proteine im
untersuchten Gewebe (auch innerhalb von Zellen) sichtbar gemacht werden
- nur eben nicht mittels Hybridisierung, sondern mit Hilfe von
Antigen-Antikörper-Reaktionen.


 Chromosomen bestehen aus einem vielfach verwickelten, aber unverzweigten Chromatinfaden - einem DNA-Molekül mit Begleitproteinen (Histone, Nicht-Histone). Histone regulieren die Verpackung und Ablesung der DNA. Multiples Aufspulen der Chromatinfäden ermöglicht platzsparende Verpackung (z.B. Metaphase der Mitose), in der Chromosomen kondensiert sind, allerdings kaum abgelesen werden können Chromosomen bestehen aus einem vielfach verwickelten, aber unverzweigten Chromatinfaden - einem DNA-Molekül mit Begleitproteinen (Histone, Nicht-Histone). Histone regulieren die Verpackung und Ablesung der DNA. Multiples Aufspulen der Chromatinfäden ermöglicht platzsparende Verpackung (z.B. Metaphase der Mitose), in der Chromosomen kondensiert sind, allerdings kaum abgelesen werden können
 Demethylierung
der DNA und Acetylierung von Histonen bewirkt eine offene Konformation
mit ablesbaren Genen, Methylierung der DNA und Deacetylierung von
Histonen geschlossene Konformation mit
"abgeschalteten" Genen. Das Epigenom
bezieht sich auf Veränderungen am Chromatin, die nicht die Basensequenz
der DNA, sondern die Realisierung (Expression) genetischer Information
betreffen Demethylierung
der DNA und Acetylierung von Histonen bewirkt eine offene Konformation
mit ablesbaren Genen, Methylierung der DNA und Deacetylierung von
Histonen geschlossene Konformation mit
"abgeschalteten" Genen. Das Epigenom
bezieht sich auf Veränderungen am Chromatin, die nicht die Basensequenz
der DNA, sondern die Realisierung (Expression) genetischer Information
betreffen
 Von einer typischen Zelle werden jeweils mehrere tausend Gene - RNA-codierende DNA-Abschnitte - ausgelesen
(transkribiert). Aktivierung von Promotorsequenzen gibt die Transkription - das Kopieren als RNA -
frei, damit beginnt die Expression (Realisierung eines Genproduktes). Die Ablesung (Transkription) von Genen erfolgt durch RNA-Polymerasen, die sich dazu an eine vor dem Gen gelegene Promotorsequenz anlagern müssen. Dazu brauchen sie DNA-bindende Proteine (Transkriptionsfaktoren). Gene können verschieden aktiviert werden, abhängig vom Muster der anregenden Faktoren. Am Transkriptionsstartpunkt müssen sich zahlreiche Proteine zusammenfinden, um die Transkription zu starten. Die Bindung regulatorischer Faktoren verändert
die Chromatinstruktur und fördert oder hemmt die Aktivität von RNA-Polymerase sowie die Bindung von Transkriptionsfaktoren Von einer typischen Zelle werden jeweils mehrere tausend Gene - RNA-codierende DNA-Abschnitte - ausgelesen
(transkribiert). Aktivierung von Promotorsequenzen gibt die Transkription - das Kopieren als RNA -
frei, damit beginnt die Expression (Realisierung eines Genproduktes). Die Ablesung (Transkription) von Genen erfolgt durch RNA-Polymerasen, die sich dazu an eine vor dem Gen gelegene Promotorsequenz anlagern müssen. Dazu brauchen sie DNA-bindende Proteine (Transkriptionsfaktoren). Gene können verschieden aktiviert werden, abhängig vom Muster der anregenden Faktoren. Am Transkriptionsstartpunkt müssen sich zahlreiche Proteine zusammenfinden, um die Transkription zu starten. Die Bindung regulatorischer Faktoren verändert
die Chromatinstruktur und fördert oder hemmt die Aktivität von RNA-Polymerase sowie die Bindung von Transkriptionsfaktoren
 Es gibt Boten- (m-), ribosomale (r-), Transfer- (t-) und weitere kleine Ribonukleinsäuren (s-RNA). Nur ~1,5% des gesamten Genoms sind proteincodierende Sequenzen; im Zellkern zunächst gebildete heteronukleäre RNA (hn-RNA) wird posttranslational bearbeitet: Entfernen von (nicht-proteincodierenden) Introns und Spleißen von (proteincodierenden) Exons; m-RNA ethält die Bauanleitung für Proteine. t-RNA
ermöglichen die
Translation (Übersetzung in die Aminosäuresprache): Sie binden einerseits ein komplementäres Basentriplet der m-RNA,
andererseits bringen sie die entsprechende Aminosäure. Proteine können posttranslational modifiziert werden Es gibt Boten- (m-), ribosomale (r-), Transfer- (t-) und weitere kleine Ribonukleinsäuren (s-RNA). Nur ~1,5% des gesamten Genoms sind proteincodierende Sequenzen; im Zellkern zunächst gebildete heteronukleäre RNA (hn-RNA) wird posttranslational bearbeitet: Entfernen von (nicht-proteincodierenden) Introns und Spleißen von (proteincodierenden) Exons; m-RNA ethält die Bauanleitung für Proteine. t-RNA
ermöglichen die
Translation (Übersetzung in die Aminosäuresprache): Sie binden einerseits ein komplementäres Basentriplet der m-RNA,
andererseits bringen sie die entsprechende Aminosäure. Proteine können posttranslational modifiziert werden
 Über 50
verschiedene Wachstumsfaktoren können die DNA-Synthese ihrer Zielzellen
aktivieren; diese benötigen dazu meist bestimmte
Muster an Wachstumsfaktoren. Mangel
an Wachstumsfaktorern bringt somatische Zellen in die G0-Phase oder
zur Apoptose. Die wichtigsten Wachstumsfaktoren sind BDNF
(brain-derived
neurotrophic factor), EGF (epidermal growth factor), TGF (transforming
growth factors), VEGF (vascular endothelial growth factors), NGF (nerve
growth factor), PDGF (platelet derived growth factors), FGF (fibroblast
growth factor), HGF / SF (hepatocyte growth factor / scatter factor),
IGF (insulin-like growth factors) Über 50
verschiedene Wachstumsfaktoren können die DNA-Synthese ihrer Zielzellen
aktivieren; diese benötigen dazu meist bestimmte
Muster an Wachstumsfaktoren. Mangel
an Wachstumsfaktorern bringt somatische Zellen in die G0-Phase oder
zur Apoptose. Die wichtigsten Wachstumsfaktoren sind BDNF
(brain-derived
neurotrophic factor), EGF (epidermal growth factor), TGF (transforming
growth factors), VEGF (vascular endothelial growth factors), NGF (nerve
growth factor), PDGF (platelet derived growth factors), FGF (fibroblast
growth factor), HGF / SF (hepatocyte growth factor / scatter factor),
IGF (insulin-like growth factors)
 Verletzungen
aktivieren Blutstillung, Kinin- und Immunsystem (Entzündung),
Wundverschluss, Gewebereparatur und Wundheilung. Integrine binden
extrazelluläre Komponenten und interagieren mit dem Zytoskelett an
fokalen Adhäsionskomplexen, Wachstumsfaktoren aktivieren die Transkription von Genen, die in ruhenden Zellen nicht abgelesen werden.
Es folgen Proliferation, Zellteilung, Differenzierung, Migration,
Apoptose; Angiogenese wird angeregt. Heilung erfolgt entlang
Leitstrukturen der extrazellulären Matrix (Organmorphologie), die dabei gespeicherte Wachstumsfaktoren freigibt Verletzungen
aktivieren Blutstillung, Kinin- und Immunsystem (Entzündung),
Wundverschluss, Gewebereparatur und Wundheilung. Integrine binden
extrazelluläre Komponenten und interagieren mit dem Zytoskelett an
fokalen Adhäsionskomplexen, Wachstumsfaktoren aktivieren die Transkription von Genen, die in ruhenden Zellen nicht abgelesen werden.
Es folgen Proliferation, Zellteilung, Differenzierung, Migration,
Apoptose; Angiogenese wird angeregt. Heilung erfolgt entlang
Leitstrukturen der extrazellulären Matrix (Organmorphologie), die dabei gespeicherte Wachstumsfaktoren freigibt
 Die Interphase besteht aus G1-Phase (mindestens 10 Stunden: Wachstum und Aktivität der Zelle), die in eine G0-Phase münden kann, ansonsten bis zum G1-Restriktionspunkt reicht, gefolgt vom G1/S-Kontrollpunkt zur Prüfung der DNA-Integrität; S-Phase (6-8 Stunden: DNA-Reduplikation); und der
~4 Stunden dauernden G2-Phase. Am abschließenden G2/M-Checkpoint können Korrekturen an der replizierten DNA erfolgen. Die Mitose
wird durch Mitogene ausgelöst und involviert Proteinkinasen
(MAP-Kinasen), welche Mitose, Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose steuern. Die Mitose besteht aus
Pro-, Meta-, Ana- und Telophase Die Interphase besteht aus G1-Phase (mindestens 10 Stunden: Wachstum und Aktivität der Zelle), die in eine G0-Phase münden kann, ansonsten bis zum G1-Restriktionspunkt reicht, gefolgt vom G1/S-Kontrollpunkt zur Prüfung der DNA-Integrität; S-Phase (6-8 Stunden: DNA-Reduplikation); und der
~4 Stunden dauernden G2-Phase. Am abschließenden G2/M-Checkpoint können Korrekturen an der replizierten DNA erfolgen. Die Mitose
wird durch Mitogene ausgelöst und involviert Proteinkinasen
(MAP-Kinasen), welche Mitose, Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose steuern. Die Mitose besteht aus
Pro-, Meta-, Ana- und Telophase
|

 Die Informationen in dieser Website basieren auf verschiedenen Quellen:
Lehrbüchern, Reviews, Originalarbeiten u.a. Sie
sollen zur Auseinandersetzung mit physiologischen Fragen, Problemen und
Erkenntnissen anregen. Soferne Referenzbereiche angegeben sind, dienen diese zur Orientierung; die Grenzen sind aus biologischen, messmethodischen und statistischen Gründen nicht absolut. Wissenschaft fragt, vermutet und interpretiert; sie ist offen, dynamisch und evolutiv. Sie strebt nach Erkenntnis, erhebt aber nicht den Anspruch, im Besitz der "Wahrheit" zu sein.
Die Informationen in dieser Website basieren auf verschiedenen Quellen:
Lehrbüchern, Reviews, Originalarbeiten u.a. Sie
sollen zur Auseinandersetzung mit physiologischen Fragen, Problemen und
Erkenntnissen anregen. Soferne Referenzbereiche angegeben sind, dienen diese zur Orientierung; die Grenzen sind aus biologischen, messmethodischen und statistischen Gründen nicht absolut. Wissenschaft fragt, vermutet und interpretiert; sie ist offen, dynamisch und evolutiv. Sie strebt nach Erkenntnis, erhebt aber nicht den Anspruch, im Besitz der "Wahrheit" zu sein.





 Gene, Proteinsynthese, Mitose, Wachstumsfaktoren, Wundheilung
Gene, Proteinsynthese, Mitose, Wachstumsfaktoren, Wundheilung




 Gene, Proteinsynthese, Mitose, Wachstumsfaktoren, Wundheilung
Gene, Proteinsynthese, Mitose, Wachstumsfaktoren, Wundheilung
 Chromosom: χρῶμα = Farbe, σῶμα = Körper ("Farbkörper")
Chromosom: χρῶμα = Farbe, σῶμα = Körper ("Farbkörper")







 Zellzyklus, Zykline, Tumorsuppressoren, Protoonkogene
Zellzyklus, Zykline, Tumorsuppressoren, Protoonkogene  Mitose
Mitose  Genom und Epigenom
Genom und Epigenom  Zellkern
Zellkern  RNA und Ribosomen, Proteinsynthese
RNA und Ribosomen, Proteinsynthese  Wachstumsfaktoren
Wachstumsfaktoren  Wundheilung
Wundheilung
 Histone
Histone  Chromosom, Chromatin, Chromatid
Chromosom, Chromatin, Chromatid  Replikation
Replikation  Gen
Gen  Codon
Codon  Exon
Exon  Expression
Expression  Promotor
Promotor  Alternatives Splicing
Alternatives Splicing  Enhancer
Enhancer  Phänotyp, Genotyp, Allel
Phänotyp, Genotyp, Allel  Homo / Heterozygotie
Homo / Heterozygotie  Zygote
Zygote  Transkriptionsfaktor
Transkriptionsfaktor  Induktion / Repression
Induktion / Repression  DNA-Methylierung
DNA-Methylierung  (Proto-) Onkogene
(Proto-) Onkogene  Mitose
Mitose  Mitogen
Mitogen  Zentrosom, Zentromer, Kinetochor
Zentrosom, Zentromer, Kinetochor  Genom
Genom  Epigenom
Epigenom  Transkription / Translation
Transkription / Translation  NF-κB
NF-κB  PCR
PCR
 Rb-Protein
Rb-Protein
 Core messages
Core messages
 Abbildung: Gene kontrollieren die Zellfunktionen
Abbildung: Gene kontrollieren die Zellfunktionen

 Abbildung: Nukleinsäuren
Abbildung: Nukleinsäuren Abbildung unten). Aneinander gereihte Nukleotide
ergeben einen Chromatinfaden, der sich zu komplexen Strukturen
zusammenknäueln (kondensieren) kann, insbesondere in Chromosomen (s. weiter unten).
Abbildung unten). Aneinander gereihte Nukleotide
ergeben einen Chromatinfaden, der sich zu komplexen Strukturen
zusammenknäueln (kondensieren) kann, insbesondere in Chromosomen (s. weiter unten).

 Abbildung: Struktur der DNA
Abbildung: Struktur der DNA
 ; diese verbinden sich längs zu Ketten, die Basen binden komplementäre Basen (A mit T, C mit G) eines
Parallelstranges.
; diese verbinden sich längs zu Ketten, die Basen binden komplementäre Basen (A mit T, C mit G) eines
Parallelstranges. 
 Abbildung: Pyrimidinbasen (oben), Purinbasen (unten)
Abbildung: Pyrimidinbasen (oben), Purinbasen (unten)
 Nukleoside bestehen aus einer Base und einer Pentose:
Nukleoside bestehen aus einer Base und einer Pentose: 
 Abbildung: Struktur eines Chromosoms
Abbildung: Struktur eines Chromosoms 
 Histone sind
basische (lysin- und argininreiche) Proteine im Chromatin, welche die
Verpackung sowie die Ablesung der DNA regulieren. Auf Histon-Oktamere
ist der DNA-Strang 1,7-fach aufgewickelt und mittels Wasserstoffbrücken fixiert; diese Struktur bildet jeweils ein Nukleosom (
Histone sind
basische (lysin- und argininreiche) Proteine im Chromatin, welche die
Verpackung sowie die Ablesung der DNA regulieren. Auf Histon-Oktamere
ist der DNA-Strang 1,7-fach aufgewickelt und mittels Wasserstoffbrücken fixiert; diese Struktur bildet jeweils ein Nukleosom (
 Abbildungen),
das 147 DNA-Basenpaare enthält. Die Energie zum Aufwickeln der DNA
stammt aus elektrostatischer Anziehung zwischen negativ geladener DNA
und positiv geladenen Histonen.
Abbildungen),
das 147 DNA-Basenpaare enthält. Die Energie zum Aufwickeln der DNA
stammt aus elektrostatischer Anziehung zwischen negativ geladener DNA
und positiv geladenen Histonen.
 Abbildung: Verpackung der DNA, Chromatin / Chromosom
Abbildung: Verpackung der DNA, Chromatin / Chromosom
 Abbildung) reduziert die Länge des Chromatinfadens auf
ein Drittel. Dazu kommen höhere Ordnungen der Kondensation; die DNA-Fäden lassen sich
dadurch auf Mikrometerlänge "zusammenpacken". An der Organisation dieser Vorgänge sind Histone und Nicht-Histon-Proteine beteiligt, jeweils zu etwa gleicher Masse wie die entsprechende DNA.
Abbildung) reduziert die Länge des Chromatinfadens auf
ein Drittel. Dazu kommen höhere Ordnungen der Kondensation; die DNA-Fäden lassen sich
dadurch auf Mikrometerlänge "zusammenpacken". An der Organisation dieser Vorgänge sind Histone und Nicht-Histon-Proteine beteiligt, jeweils zu etwa gleicher Masse wie die entsprechende DNA.  Chromatin ist der gesamte Komplex aus DNA, Histon- und Nicht-Histon-Proteinen, aus dem Chromosomen aufgebaut sind. Jedes Chromatid besteht aus einem DNA-Doppelstrang sowie zugehörigen Chromatin-Proteinen. Je nach Phase
des Zellzyklus kann ein Chromosom aus einem oder mehreren Chromatiden
bestehen.
Chromatin ist der gesamte Komplex aus DNA, Histon- und Nicht-Histon-Proteinen, aus dem Chromosomen aufgebaut sind. Jedes Chromatid besteht aus einem DNA-Doppelstrang sowie zugehörigen Chromatin-Proteinen. Je nach Phase
des Zellzyklus kann ein Chromosom aus einem oder mehreren Chromatiden
bestehen. Replikation (Reduplikation, DNA / RNA replication) ist
die Vervielfältigung von Erbinformation in Zellen (oder Viren). Zur
Replikation von DNA bedarf es der Mitwirkung verschiedener Proteine:
Replikation (Reduplikation, DNA / RNA replication) ist
die Vervielfältigung von Erbinformation in Zellen (oder Viren). Zur
Replikation von DNA bedarf es der Mitwirkung verschiedener Proteine: Topoisomerasen
starten den Vorgang des Abwickelns ("Entdröselns") der DNA durch
Aufbrechen eines Stranges und dadurch Entspannung der Helixstruktur
Topoisomerasen
starten den Vorgang des Abwickelns ("Entdröselns") der DNA durch
Aufbrechen eines Stranges und dadurch Entspannung der Helixstruktur Helikasen glätten die Doppelhelix nach Einwirken von Topoisomerasen an der Replikationsgabel
Helikasen glätten die Doppelhelix nach Einwirken von Topoisomerasen an der Replikationsgabel Einzelstrang-DNA bindende Proteine (single-strand-DNA binding proteins) erhalten die Stabilität der Replikationsgabel (Einzelstrang-DNA ist sehr empfindlich und leicht abbaubar)
Einzelstrang-DNA bindende Proteine (single-strand-DNA binding proteins) erhalten die Stabilität der Replikationsgabel (Einzelstrang-DNA ist sehr empfindlich und leicht abbaubar) Primasen befestigen kurze komplementäre RNA-Sequenzen (Primer) am DNA-Einzelstrang der Replikationsgabel
Primasen befestigen kurze komplementäre RNA-Sequenzen (Primer) am DNA-Einzelstrang der Replikationsgabel DNA-Polymerasen erzeugen entweder eine DNA-Kopie
(DNA-Polymerasen) oder RNA (RNA-Polymerasen). RNA-Polymerase I (Pol I)
transkribiert ribosomale (r) RNA, RNA-Polymerase II (Pol II) Boten (m)
RNA und die meisten kleinen nukleären (sn) RNA, RNA-Polymerase III (Pol
III) transfer (t) RNA und einige snRNA (vgl.
DNA-Polymerasen erzeugen entweder eine DNA-Kopie
(DNA-Polymerasen) oder RNA (RNA-Polymerasen). RNA-Polymerase I (Pol I)
transkribiert ribosomale (r) RNA, RNA-Polymerase II (Pol II) Boten (m)
RNA und die meisten kleinen nukleären (sn) RNA, RNA-Polymerase III (Pol
III) transfer (t) RNA und einige snRNA (vgl.  unten):
unten): DNA-Ligasen schweißen Knickstellen zwischen neusynthetisierter DNA zusammen.
DNA-Ligasen schweißen Knickstellen zwischen neusynthetisierter DNA zusammen.
 Gene
Gene
 sind DNA-Abschnitte, die den Bauplan einer mRNA
(messenger ribonucleic acid, Botenribonukleinsäure) und damit den eines Proteins codieren. Gene, die "abgeschrieben"
(transkribiert) werden, heißen Transkriptionseinheiten (transcription units).
sind DNA-Abschnitte, die den Bauplan einer mRNA
(messenger ribonucleic acid, Botenribonukleinsäure) und damit den eines Proteins codieren. Gene, die "abgeschrieben"
(transkribiert) werden, heißen Transkriptionseinheiten (transcription units).  Abbildung). Gene nehmen in bestimmten Chromosomen bestimmte Positionen ein - sogenannte Genloci (Einzahl: Genlocus).
Abbildung). Gene nehmen in bestimmten Chromosomen bestimmte Positionen ein - sogenannte Genloci (Einzahl: Genlocus).
 Abbildung: Gen und Genprodukte
Abbildung: Gen und Genprodukte Abbildung). Das Gen-Ende ist durch ein Polyadenylierungssignal
gekennzeichnet (meist AATAAA).
Abbildung). Das Gen-Ende ist durch ein Polyadenylierungssignal
gekennzeichnet (meist AATAAA). Ein Codon ist ein kurzes Stück mRNA (ein Basentriplett), das einen Synthesebeginn (Start-Codon), eine Aminosäure oder einen Transkriptionsabbruch (Stop-Codon) codiert. tRNA-Moleküle enthalten komplimentäre Codons (Anticodons) und bringen die jeweils "passende" Aminosäure ein.
Ein Codon ist ein kurzes Stück mRNA (ein Basentriplett), das einen Synthesebeginn (Start-Codon), eine Aminosäure oder einen Transkriptionsabbruch (Stop-Codon) codiert. tRNA-Moleküle enthalten komplimentäre Codons (Anticodons) und bringen die jeweils "passende" Aminosäure ein. Exons
Exons  sind proteincodierende RNA-Sequenzen. Spleißen (splicing) ist das Entfernen von Introns
sind proteincodierende RNA-Sequenzen. Spleißen (splicing) ist das Entfernen von Introns  - nicht-codierenden RNA-Sequenzen - aus Prä-mRNA. (Als Exom bezeichnet man die proteincodierende Region des Genoms insgesamt - nicht zu verwechseln mit Exon.)
- nicht-codierenden RNA-Sequenzen - aus Prä-mRNA. (Als Exom bezeichnet man die proteincodierende Region des Genoms insgesamt - nicht zu verwechseln mit Exon.)  Genexpression (Exprimierung, Expression) ist
der Vorgang, der genetische Information in die Synthese funktionaler
Genprodukte umsetzt (
Genexpression (Exprimierung, Expression) ist
der Vorgang, der genetische Information in die Synthese funktionaler
Genprodukte umsetzt ( Abbildung unten). Man sagt, die Zelle exprimiert einen Stoff (z.B. Protein). Tut sie das immer, ist der Stoff ein konstitutiver Bestandteil, betreffende (konstitutive) Gene werden ständig exprimiert (und kümmern sich um grundlegende Zellfunktionen: housekeeping genes). Fakultative Gene werden hingegen nur bei Bedarf (z.B. Zellteilung) bzw. durch Anregung der Zelle - also situationsabhängig - exprimiert (inducible genes).
Abbildung unten). Man sagt, die Zelle exprimiert einen Stoff (z.B. Protein). Tut sie das immer, ist der Stoff ein konstitutiver Bestandteil, betreffende (konstitutive) Gene werden ständig exprimiert (und kümmern sich um grundlegende Zellfunktionen: housekeeping genes). Fakultative Gene werden hingegen nur bei Bedarf (z.B. Zellteilung) bzw. durch Anregung der Zelle - also situationsabhängig - exprimiert (inducible genes). Ein Promotor (promoter) ist
eine - unmittelbar vor dem 5'-Ende der betreffenden Startstelle der
Transkription gelegene - DNA-Sequenz, die notwendig ist, um ein Gen zu
exprimieren und welche die Richtung der Transkription bestimmt. Viele
Promotoren enthalten eine TATA-Box, eine thymin- und adeninreichen Sequenz, meist etwa 30 Basenpaare vor dem Ort des Transkriptionsbeginns gelegen. Promotoren sind Kombinationen aus Nukleotid-Sequenzen, meist unmittelbar vor dem betreffenden Gen (upstream) gelegen, an die RNA-Polymerase bindet und die Gen-Expression startet.
Ein Promotor (promoter) ist
eine - unmittelbar vor dem 5'-Ende der betreffenden Startstelle der
Transkription gelegene - DNA-Sequenz, die notwendig ist, um ein Gen zu
exprimieren und welche die Richtung der Transkription bestimmt. Viele
Promotoren enthalten eine TATA-Box, eine thymin- und adeninreichen Sequenz, meist etwa 30 Basenpaare vor dem Ort des Transkriptionsbeginns gelegen. Promotoren sind Kombinationen aus Nukleotid-Sequenzen, meist unmittelbar vor dem betreffenden Gen (upstream) gelegen, an die RNA-Polymerase bindet und die Gen-Expression startet. Alternatives (RNA-) splicing ist eine Variante der Umsetzung eines "Vorläufer"-mRNA-Stanges (precursor messenger RNA, pre-mRNA) zu "reifer" RNA (mature messenger RNA, mRNA). Es kann
von so gut wie jedem Gen ausgehen. So können einige Exons eines Gens in die endgültige mRNA eingeschlossen sein - oder nicht -, was zur Bildung
unterschiedlicher Proteine führt.
Alternatives (RNA-) splicing ist eine Variante der Umsetzung eines "Vorläufer"-mRNA-Stanges (precursor messenger RNA, pre-mRNA) zu "reifer" RNA (mature messenger RNA, mRNA). Es kann
von so gut wie jedem Gen ausgehen. So können einige Exons eines Gens in die endgültige mRNA eingeschlossen sein - oder nicht -, was zur Bildung
unterschiedlicher Proteine führt.  Abbildung). TFIID enthält ein TATA-bindendes Protein.
Abbildung). TFIID enthält ein TATA-bindendes Protein.
 Abbildung: Regulatorische Sequenzen und Promotor
Abbildung: Regulatorische Sequenzen und Promotor Abbildung oben
Abbildung oben
 Enhancer sind
regulatorische DNA-Elemente, die sich in einigem Abstand (bis zu 1
Megabasen upstream oder downstream) von einem Gen befinden, dessen
Expression kontrolliert werden soll. Durch - mittels eines als Cohesin bezeichneten Proteinkomplexes stabilisierte - Schleifenbildung (DNA-looping) (
Enhancer sind
regulatorische DNA-Elemente, die sich in einigem Abstand (bis zu 1
Megabasen upstream oder downstream) von einem Gen befinden, dessen
Expression kontrolliert werden soll. Durch - mittels eines als Cohesin bezeichneten Proteinkomplexes stabilisierte - Schleifenbildung (DNA-looping) ( Abbildung)
kommen sie mit ihrem Zielgen in direkten Kontakt. Enhancer bewirken den
Großteil an gewebe- bzw. zellspezifischer Genexpression, und ein
bestimmtes Gen kann zahlreiche Enhancer nutzen, die jeweils einen
bestimmten Aspekt der gewebespezifischen Genexpression steuern.
Abbildung)
kommen sie mit ihrem Zielgen in direkten Kontakt. Enhancer bewirken den
Großteil an gewebe- bzw. zellspezifischer Genexpression, und ein
bestimmtes Gen kann zahlreiche Enhancer nutzen, die jeweils einen
bestimmten Aspekt der gewebespezifischen Genexpression steuern.
 Abbildung: Interaktion verschiedener Faktoren am Transkriptionskomplex
Abbildung: Interaktion verschiedener Faktoren am Transkriptionskomplex
 Als Phänotyp
Als Phänotyp  bezeichnet man die (beobachtbaren) Eigenschaften und Charakteristika eines Organismus. Der Genotyp ist seine Ausstattung mit Genen (der Begriff wird meist gebraucht in Bezug auf bestimmte Allele. Ein Allel ist eine vererbte
genetische Variante einer bestimmten DNA-Sequenz innerhalb des Genoms.
Beispielsweise sind interindividuelle Unterschiede bezüglich
Blutgruppe, Augenfarbe etc. durch Allele entsprechender Gene bedingt; diese sitzen an definierten Orten bestimmter Chromosome.
bezeichnet man die (beobachtbaren) Eigenschaften und Charakteristika eines Organismus. Der Genotyp ist seine Ausstattung mit Genen (der Begriff wird meist gebraucht in Bezug auf bestimmte Allele. Ein Allel ist eine vererbte
genetische Variante einer bestimmten DNA-Sequenz innerhalb des Genoms.
Beispielsweise sind interindividuelle Unterschiede bezüglich
Blutgruppe, Augenfarbe etc. durch Allele entsprechender Gene bedingt; diese sitzen an definierten Orten bestimmter Chromosome.  Bezüglich eines bestimmten Gens homozygot ist ein
Organismus, wenn beide Allele des betreffenden Genlocus (eines von der Mutter, eines vom Vater) eines homologen Chromosomenpaares die gleiche Nukleotidsequenz aufweisen (genetisch identisch sind). Sind die Nukleotidsequenzen der Allele
unterschiedlich, ist der Organismus bezüglich dieses Gens heterozygot
(auch wenn sich die Gene nur in einem einzigen Nukleotid
unterscheiden). Gene können in zwei oder mehr allelen Zuständen
vorliegen (20-30% der Fälle), woraus sich in der Population eine große genetische Vielfalt ergibt.
Bezüglich eines bestimmten Gens homozygot ist ein
Organismus, wenn beide Allele des betreffenden Genlocus (eines von der Mutter, eines vom Vater) eines homologen Chromosomenpaares die gleiche Nukleotidsequenz aufweisen (genetisch identisch sind). Sind die Nukleotidsequenzen der Allele
unterschiedlich, ist der Organismus bezüglich dieses Gens heterozygot
(auch wenn sich die Gene nur in einem einzigen Nukleotid
unterscheiden). Gene können in zwei oder mehr allelen Zuständen
vorliegen (20-30% der Fälle), woraus sich in der Population eine große genetische Vielfalt ergibt. Unter einer Zygote
Unter einer Zygote  versteht man eine (diploide) Zelle, die durch Fertilisation aus zwei (hapoliden) Gameten (einer
Eizelle und ein Spermium) entstanden ist. Bei der Fusionierung der
beiden Chromosomensätze komplettiert die Eizelle ihre Meiose 2. Die
Zygote bereitet sich anschließend auf die erste mitotische Teilung vor
und ist vorübergehend 4n-dipoid. Durch mehrfache Mitose wird das 2- und
4-Zellen-Stadium durchlaufen, anschließend entsteht eine Morula und
durch Flüssigkeitseinlagerung eine Blastula (Blastogenese).
versteht man eine (diploide) Zelle, die durch Fertilisation aus zwei (hapoliden) Gameten (einer
Eizelle und ein Spermium) entstanden ist. Bei der Fusionierung der
beiden Chromosomensätze komplettiert die Eizelle ihre Meiose 2. Die
Zygote bereitet sich anschließend auf die erste mitotische Teilung vor
und ist vorübergehend 4n-dipoid. Durch mehrfache Mitose wird das 2- und
4-Zellen-Stadium durchlaufen, anschließend entsteht eine Morula und
durch Flüssigkeitseinlagerung eine Blastula (Blastogenese). Transkriptionsfaktoren sind
Proteine, die sich meist an spezifische DNA-Sequenzen anlagern und die
Expression bestimmter Zielgene regulieren. Ihre Bindungsstellen nennt
man regulatorische Sequenzen bzw. Elemente. Als Cis-Element bezeichnet man einen DNA-Abschnitt, der die Ablesung eines Gens auf dem DNA-Faden verstärkt (Enhancer) oder abschwächt (Silencer). Promoter sind Cis-Elemente. An Enhancersequenzen der DNA binden als Aktivatoren bezeichnete Transkriptionsfaktoren. Andere Transkriptionsfaktoren wirken als Repressoren, sie binden an Silencersequenzen.
Transkriptionsfaktoren sind
Proteine, die sich meist an spezifische DNA-Sequenzen anlagern und die
Expression bestimmter Zielgene regulieren. Ihre Bindungsstellen nennt
man regulatorische Sequenzen bzw. Elemente. Als Cis-Element bezeichnet man einen DNA-Abschnitt, der die Ablesung eines Gens auf dem DNA-Faden verstärkt (Enhancer) oder abschwächt (Silencer). Promoter sind Cis-Elemente. An Enhancersequenzen der DNA binden als Aktivatoren bezeichnete Transkriptionsfaktoren. Andere Transkriptionsfaktoren wirken als Repressoren, sie binden an Silencersequenzen. andauernd hergestellte für die permanente Grundversorgung der Zelle mit RNA, die an den unmittelbar vor der codierenden DNA-Region liegenden Kernpromotor binden (basale Transkriptionsfaktoren),
andauernd hergestellte für die permanente Grundversorgung der Zelle mit RNA, die an den unmittelbar vor der codierenden DNA-Region liegenden Kernpromotor binden (basale Transkriptionsfaktoren), auf regulierende weiter proximal gelegene DNA-Steuerelemente zugreifende stromaufwärts bindende (s. Abbildung),
auf regulierende weiter proximal gelegene DNA-Steuerelemente zugreifende stromaufwärts bindende (s. Abbildung), von außen oder durch den Zellzyklus gesteuerte induzierbare Transkriptionsfaktoren, z.B. durch Hormone - wie z,B. Steroide, Katecholamine - und deren Signalstoffe (second messenger) induzierte, oder das P53-Protein - diese Faktoren werden bedarfsmäßig aktiviert, müssen also je nach Erfordernissen "eingeschaltet" werden.
von außen oder durch den Zellzyklus gesteuerte induzierbare Transkriptionsfaktoren, z.B. durch Hormone - wie z,B. Steroide, Katecholamine - und deren Signalstoffe (second messenger) induzierte, oder das P53-Protein - diese Faktoren werden bedarfsmäßig aktiviert, müssen also je nach Erfordernissen "eingeschaltet" werden. AP-1 (Activator Protein 1), steuert mit Differenzierung, Teilung und Apoptose der Zelle befasste Gene
AP-1 (Activator Protein 1), steuert mit Differenzierung, Teilung und Apoptose der Zelle befasste Gene ATF / CREB-Familie, eine Gruppe von Transkriptionsfaktoren, bestehend aus ATF (Activating transcription factors) und CREB, verfügen über eine DNA-Bindungsdomäne vom Leuzin-Zipper-Typ, die Proteine dimerisiert
ATF / CREB-Familie, eine Gruppe von Transkriptionsfaktoren, bestehend aus ATF (Activating transcription factors) und CREB, verfügen über eine DNA-Bindungsdomäne vom Leuzin-Zipper-Typ, die Proteine dimerisiert Hitzeschockfaktoren, diese regulieren die Expression von Hitzeschockproteinen
Hitzeschockfaktoren, diese regulieren die Expression von Hitzeschockproteinen IRF, Interferon regulatory factors, die Transkription von Interferonen regulierende Proteinediese regulieren die Expression von Hitzeschockproteinen
IRF, Interferon regulatory factors, die Transkription von Interferonen regulierende Proteinediese regulieren die Expression von Hitzeschockproteinen NF-κB (NF-kappa B, nuclear factor kappa-light-chain-enhancer of activated B cells)
NF-κB (NF-kappa B, nuclear factor kappa-light-chain-enhancer of activated B cells) SP-1 (Specificity Protein 1), ein Zinkfinger-Transkriptionsfaktor, der in zahlreiche Vorgänge (Differenzierung, Wachstum, Immunantworten etc) involviert ist
SP-1 (Specificity Protein 1), ein Zinkfinger-Transkriptionsfaktor, der in zahlreiche Vorgänge (Differenzierung, Wachstum, Immunantworten etc) involviert ist NF-κB ist
ein in fast allen Zellen aktiver Proteinkomplex, besonders in
proliferierenden Zellen, z.B. Immunzellen (Entwicklung, Entzündung). NF-κB bindet an eine Sequenz von ~10 Basenpaaren (κB-Motiv) und verstärkt meist die Transkription. Es kann die Aktivität von T-Zellen und NK-Zellen steigern, wenn das angeborene Immunsystem gereizt wird - über erhöhte Spiegel proinflammatorischer Zytokine
NF-κB ist
ein in fast allen Zellen aktiver Proteinkomplex, besonders in
proliferierenden Zellen, z.B. Immunzellen (Entwicklung, Entzündung). NF-κB bindet an eine Sequenz von ~10 Basenpaaren (κB-Motiv) und verstärkt meist die Transkription. Es kann die Aktivität von T-Zellen und NK-Zellen steigern, wenn das angeborene Immunsystem gereizt wird - über erhöhte Spiegel proinflammatorischer Zytokine Abbildung):
Abbildung):
 Abbildung: Phasen der Genexpression
Abbildung: Phasen der Genexpression Induktion ist
die Ermöglichung einer Genexpression (z.B. durch ein
Stoffwechselprodukt), indem ein Aktivator (Definition s. oben) ein- bzw. Repressor
(Definition s. oben) ausgeschaltet wird; Repression bedeutet, dass umgekehrt eine Genexpression verhindert wird (indem ein Repressor aktiviert bzw. ein Aktivator ausgeschaltet wird).
Induktion ist
die Ermöglichung einer Genexpression (z.B. durch ein
Stoffwechselprodukt), indem ein Aktivator (Definition s. oben) ein- bzw. Repressor
(Definition s. oben) ausgeschaltet wird; Repression bedeutet, dass umgekehrt eine Genexpression verhindert wird (indem ein Repressor aktiviert bzw. ein Aktivator ausgeschaltet wird). von Erbanlagen im klassischen Sinne (Genausstattung: Genom),
von Erbanlagen im klassischen Sinne (Genausstattung: Genom), Umweltfaktoren
(Wasser-, Elektrolyt-, Atemgas-, Nährstoff-, Vitamin-,
Spurenelementangebot, Temperaturprofil, Strahlung, Toxine, ..) - diese
können auch epigenetisch wirksam werden, indem sie z.B. das
Methylierungsmuster bestimmter Gene beeinflussen -, sowie
Umweltfaktoren
(Wasser-, Elektrolyt-, Atemgas-, Nährstoff-, Vitamin-,
Spurenelementangebot, Temperaturprofil, Strahlung, Toxine, ..) - diese
können auch epigenetisch wirksam werden, indem sie z.B. das
Methylierungsmuster bestimmter Gene beeinflussen -, sowie anderen Faktoren, wie mobilen
genetischen Elementen (Retrotransposonen),
die Kopien von sich an zufälligen Stellen ins
Genom einfügen können.
anderen Faktoren, wie mobilen
genetischen Elementen (Retrotransposonen),
die Kopien von sich an zufälligen Stellen ins
Genom einfügen können. DNA-Methylierung ist das Anfügen von Methylgruppen (CH3)
an ein DNA-Molekül (einen Chromatinfaden) mittels
DNA-Methyltransferasen. Dabei wird nicht die betreffende DNA-Sequenz
geändert, sondern ihre Ablesbarkeit bzw. Aktivität: DNA-Sequenzen
können ablesbar oder "maskiert" sein. Methylierung
kann Gene und/oder ihre Regulatoren (Enhancer, Promoter..)
inaktivieren. DNA-Methylierung ist der wichtigste Vorgang im Rahmen epigenetischer Veränderungen.
DNA-Methylierung ist das Anfügen von Methylgruppen (CH3)
an ein DNA-Molekül (einen Chromatinfaden) mittels
DNA-Methyltransferasen. Dabei wird nicht die betreffende DNA-Sequenz
geändert, sondern ihre Ablesbarkeit bzw. Aktivität: DNA-Sequenzen
können ablesbar oder "maskiert" sein. Methylierung
kann Gene und/oder ihre Regulatoren (Enhancer, Promoter..)
inaktivieren. DNA-Methylierung ist der wichtigste Vorgang im Rahmen epigenetischer Veränderungen.  der “Arbeitsfunktion” (Interphase),
in der Gene exprimiert (abgelesen und "übersetzt") werden (Synthese von RNA und Proteinen - die
Chromatinfäden liegen im Zellkern als mehr oder weniger entfaltete, gut
ablesbare Fäden
vor) und (vor der nächsten Mitose) die Chromatiden repliziert werden (was
zur Bildung von Schwesterchromatidenpaaren führt -
Replikationsgeschwindigkeit: Bei Bakterien bis zu ~1000, in
eukaryotischen Zellen ~50 Nukleotide pro Sekunde), oder
der “Arbeitsfunktion” (Interphase),
in der Gene exprimiert (abgelesen und "übersetzt") werden (Synthese von RNA und Proteinen - die
Chromatinfäden liegen im Zellkern als mehr oder weniger entfaltete, gut
ablesbare Fäden
vor) und (vor der nächsten Mitose) die Chromatiden repliziert werden (was
zur Bildung von Schwesterchromatidenpaaren führt -
Replikationsgeschwindigkeit: Bei Bakterien bis zu ~1000, in
eukaryotischen Zellen ~50 Nukleotide pro Sekunde), oder  der Bildung neuer Zellen (Kondensation der Chromatiden zu Chromosomen, Voraussetzung für die Mitose).
der Bildung neuer Zellen (Kondensation der Chromatiden zu Chromosomen, Voraussetzung für die Mitose). Abbildung) in die G1, S- und G2-Phase:
Abbildung) in die G1, S- und G2-Phase: G1-Phase
(Gap 1): Dies ist der "Arbeitszustand", in dem die (diploide) Zelle ihre
Dienste für den Organismus leistet. Ihre Mindestdauer beträgt ~10
Stunden (die Zelle wächst hier auch und kann sich nicht sofort wieder
teilen). Die Dauer hängt von den Bedingungen ab, in denen sich die Zelle wiederfindet. Signale durch Mitogene
heben die Wirkung intrazellulär hemmender Kontrollfaktoren auf; die
Zelle muss aber auch entscheiden, ob genügend Ressourcen für weitere
Mitosen zur Verfügung stehen - unabhängig von der Anwesenheit mitogener
Signale.
G1-Phase
(Gap 1): Dies ist der "Arbeitszustand", in dem die (diploide) Zelle ihre
Dienste für den Organismus leistet. Ihre Mindestdauer beträgt ~10
Stunden (die Zelle wächst hier auch und kann sich nicht sofort wieder
teilen). Die Dauer hängt von den Bedingungen ab, in denen sich die Zelle wiederfindet. Signale durch Mitogene
heben die Wirkung intrazellulär hemmender Kontrollfaktoren auf; die
Zelle muss aber auch entscheiden, ob genügend Ressourcen für weitere
Mitosen zur Verfügung stehen - unabhängig von der Anwesenheit mitogener
Signale.
 Abbildung: Phasen des Zellzyklus
Abbildung: Phasen des Zellzyklus
 Zahlreiche Zellen treten postmitotisch in die G0-Phase ein (diese wird auch als Ruhephase
(resting stage) bezeichnet - was sich auf die Mitoseaktivität bezieht, die Zellen selbst können in der G0-Phase
hochaktiv sein, z.B. wachsen, sich bewegen, migrieren, Stoffe aufbauen
etc). In diesem modifizierten G1-Stadium verharren sie entweder
Zahlreiche Zellen treten postmitotisch in die G0-Phase ein (diese wird auch als Ruhephase
(resting stage) bezeichnet - was sich auf die Mitoseaktivität bezieht, die Zellen selbst können in der G0-Phase
hochaktiv sein, z.B. wachsen, sich bewegen, migrieren, Stoffe aufbauen
etc). In diesem modifizierten G1-Stadium verharren sie entweder
 vorübergehend (wenn auch u.U. sehr lange,
z.B. Stammzellen). Aus dieser Phase können (bei G1)
Zellen in den Zyklus jederzeit wieder eintreten, wenn sie entsprechend
stimuliert werden (z.B. durch Wachstumsfaktoren). Für gealterte Zellen
gilt das nicht. Auch vollständig ausdifferenzierte Zellen verbleiben
normalerweise in G0;
vorübergehend (wenn auch u.U. sehr lange,
z.B. Stammzellen). Aus dieser Phase können (bei G1)
Zellen in den Zyklus jederzeit wieder eintreten, wenn sie entsprechend
stimuliert werden (z.B. durch Wachstumsfaktoren). Für gealterte Zellen
gilt das nicht. Auch vollständig ausdifferenzierte Zellen verbleiben
normalerweise in G0;  oder permanent (das trifft auf die meisten Nervenzellen sowie auf das Myokard zu,
die dauerhaft spezialisiert bleiben).
oder permanent (das trifft auf die meisten Nervenzellen sowie auf das Myokard zu,
die dauerhaft spezialisiert bleiben).  In der G1- und G0-Phase vollbringt das Genom den Großteil seiner Arbeit (Transkription).
In der G1- und G0-Phase vollbringt das Genom den Großteil seiner Arbeit (Transkription). In der S-Phase
(DNA-Synthesephase: Reduplikation) kopiert (verdoppelt) die Zelle ihr Erbmaterial
(DNA-Reduplikation).
In der frühen S-Phase befindet sich in jedem Chromosom eine DNA-Doppelhelix (Chromosomen 2N, DNA 2C), in der späten S-Phase - nach der Reduplikation - verfügt jedes Chromosom über zwei DNA-Doppelhelices (Chromosomen 2N, DNA 4C - vgl. dort). Die beiden Tochterzellen verfügen nach der Zellteilung wieder über 2N / 2C.
In der S-Phase
(DNA-Synthesephase: Reduplikation) kopiert (verdoppelt) die Zelle ihr Erbmaterial
(DNA-Reduplikation).
In der frühen S-Phase befindet sich in jedem Chromosom eine DNA-Doppelhelix (Chromosomen 2N, DNA 2C), in der späten S-Phase - nach der Reduplikation - verfügt jedes Chromosom über zwei DNA-Doppelhelices (Chromosomen 2N, DNA 4C - vgl. dort). Die beiden Tochterzellen verfügen nach der Zellteilung wieder über 2N / 2C. (das innere Kinetochor ist fest an die DNA befestigt, das äußere interagiert mit Mikrotubuli des Spindelapparates).
(das innere Kinetochor ist fest an die DNA befestigt, das äußere interagiert mit Mikrotubuli des Spindelapparates).  Die beiden Schwesterchromatiden bleiben dann eng aneinandergeheftet und
werden zur Bildung von Mitosechromosomen weiter kondensiert.
Die beiden Schwesterchromatiden bleiben dann eng aneinandergeheftet und
werden zur Bildung von Mitosechromosomen weiter kondensiert.  G2-Phase (Gap 2) dient der finalen Vorbereitung auf die Mitose. In dieser
~4 Stunden dauernden Zeit der "Kontemplation" kontrolliert die Zelle das Ergebnis der
vorausgegangenen Reduplikationsvorgänge (DNA korrekt verdoppelt?). Am abschließenden G2/M-Checkpoint (G2/M-Übergang) werden eventuell notwendige Korrekturen
an der replizierten DNA vorgenommen (gesamte DNA verdoppelt?). Fällt
die Überprüfung zufriedenstellend aus, erfolgt der Eintritt in die
Mitose; sind bei der Replikation
zu viele Fehler passiert, wird
G2-Phase (Gap 2) dient der finalen Vorbereitung auf die Mitose. In dieser
~4 Stunden dauernden Zeit der "Kontemplation" kontrolliert die Zelle das Ergebnis der
vorausgegangenen Reduplikationsvorgänge (DNA korrekt verdoppelt?). Am abschließenden G2/M-Checkpoint (G2/M-Übergang) werden eventuell notwendige Korrekturen
an der replizierten DNA vorgenommen (gesamte DNA verdoppelt?). Fällt
die Überprüfung zufriedenstellend aus, erfolgt der Eintritt in die
Mitose; sind bei der Replikation
zu viele Fehler passiert, wird  der Vorgang verzögert, um die entsprechenden Reparaturen vorzunehmen,
der Vorgang verzögert, um die entsprechenden Reparaturen vorzunehmen,  die Zelle mitotisch stillgestellt ("Seneszenz", vor allem durch Wirkung des
P53-Proteins, s. unten), oder
die Zelle mitotisch stillgestellt ("Seneszenz", vor allem durch Wirkung des
P53-Proteins, s. unten), oder  die Zelle der Apoptose zugeführt.
die Zelle der Apoptose zugeführt.  Details zum Ablauf der Mitose s. weiter unten
Details zum Ablauf der Mitose s. weiter unten
 Abbildung: Kontrollpunkte (Restriktionspunkte) des Zellzyklus
Abbildung: Kontrollpunkte (Restriktionspunkte) des Zellzyklus
 Abbildung): G1/S, G2/M,
und ein Metaphase- zu- Anaphase- Kontrollpunkt. An diesen
Übergangsstellen können allfällig notwendige Reparaturen vorgenommen
werden.
Abbildung): G1/S, G2/M,
und ein Metaphase- zu- Anaphase- Kontrollpunkt. An diesen
Übergangsstellen können allfällig notwendige Reparaturen vorgenommen
werden. Kontrollpunkt 1. Am G1-Restriktionspunkt
(G1/S-Übergang) fällt die Entscheidung, ob die Zelle in die Synthesephase eintreten kann. Dieser Punkt ist komplex reguliert, er liegt an einer besonders entscheidenden Stelle (ist die
Zelle zur Reproduktion befähigt? Liegen Signale vor, welche die Mitose begünstigen?). Lautet die Antwort "ja", sind die weiteren Vorgänge
von Außenfaktoren unabhängig, und es erfolgt die DNA-Reduplikation. Dazu ist z.B. die Anwesenheit
bestimmter extrazellulärer Signale erforderlich (Wachstums- und
Adhäsionsfaktoren). Wachstumsfaktoren müssen an ihre Rezeptoren binden, Proteine in der Zelle phosphoryliert
werden und Proliferationsvorgänge starten.
Kontrollpunkt 1. Am G1-Restriktionspunkt
(G1/S-Übergang) fällt die Entscheidung, ob die Zelle in die Synthesephase eintreten kann. Dieser Punkt ist komplex reguliert, er liegt an einer besonders entscheidenden Stelle (ist die
Zelle zur Reproduktion befähigt? Liegen Signale vor, welche die Mitose begünstigen?). Lautet die Antwort "ja", sind die weiteren Vorgänge
von Außenfaktoren unabhängig, und es erfolgt die DNA-Reduplikation. Dazu ist z.B. die Anwesenheit
bestimmter extrazellulärer Signale erforderlich (Wachstums- und
Adhäsionsfaktoren). Wachstumsfaktoren müssen an ihre Rezeptoren binden, Proteine in der Zelle phosphoryliert
werden und Proliferationsvorgänge starten.  Abbildung):
Abbildung):
 Abbildung: Aktivierung einer cyclinabhängigen Kinase (cdk)
Abbildung: Aktivierung einer cyclinabhängigen Kinase (cdk)
 Cyclin D-CDK4 / CDK6 steuert Vorgänge der frühen G1-Phase
Cyclin D-CDK4 / CDK6 steuert Vorgänge der frühen G1-Phase Cyclin D/E - CDK2 triggert die S-Phase
Cyclin D/E - CDK2 triggert die S-Phase Cyclin A - CDK1 / CDK2 kümmert sich um den Abschluss der S-Phase
Cyclin A - CDK1 / CDK2 kümmert sich um den Abschluss der S-Phase Für die Mitose ist Cyclin B - CDK1 essentiell
Für die Mitose ist Cyclin B - CDK1 essentiell Die Interphase ist Cyclin A - CDK1 / CDK2 -gesteuert
Die Interphase ist Cyclin A - CDK1 / CDK2 -gesteuert Abbildung).
Abbildung).
 Abbildung: Zellzyklus und Rolle von cdk-Komplexen
Abbildung: Zellzyklus und Rolle von cdk-Komplexen
 Onkogene
sind Gene, die meist Wachstumsfaktoren, deren Rezeptoren,
Signaltransducer oder Transkriptionsfaktoren codieren. Mutieren sie,
werden sie oft überaktiv oder resistent gegenüber Hemmsignalen; das
kann Krebsbildung zur Folge haben. Die meisten Onkogene beginnen diese
Entwicklung als Protoonkogene;
diese unterstützen das Zellwachstum und beteiligen sich an der Hemmung einer
Apoptose. Mutieren Protoonkogene unter Viren-,
Strahlungs-, oder Mutageneinfluss, kann sich ihre Funktion verstärken (gain-of-function mutation). Dies kann zu Tumorentstehung führen; die Mutation eines einzigen Gens reicht schon dazu aus.
Onkogene
sind Gene, die meist Wachstumsfaktoren, deren Rezeptoren,
Signaltransducer oder Transkriptionsfaktoren codieren. Mutieren sie,
werden sie oft überaktiv oder resistent gegenüber Hemmsignalen; das
kann Krebsbildung zur Folge haben. Die meisten Onkogene beginnen diese
Entwicklung als Protoonkogene;
diese unterstützen das Zellwachstum und beteiligen sich an der Hemmung einer
Apoptose. Mutieren Protoonkogene unter Viren-,
Strahlungs-, oder Mutageneinfluss, kann sich ihre Funktion verstärken (gain-of-function mutation). Dies kann zu Tumorentstehung führen; die Mutation eines einzigen Gens reicht schon dazu aus. Kontrollpunkt 2 (G2/M-Übergang). Der G2/M-Kontrollpunkt ist weniger komplex reguliert als G1/S und stellt eine Art Notbremse
dar, etwa für den Fall, dass die Replikation fehlerhaft verlaufen ist (s. oben). Die Aktivität der Kinasen schwankt mit dem Zellzyklus. So ist am G2/M-Übergang
die cdk-Aktivität hoch, und Proteine, welche die
Vorgänge der frühen Mitose steuern, werden vermehrt aktiviert (Abbau der Kernhülle, Kondensation
der Chromosomen, Aufbau der Spindeltubuli).
Kontrollpunkt 2 (G2/M-Übergang). Der G2/M-Kontrollpunkt ist weniger komplex reguliert als G1/S und stellt eine Art Notbremse
dar, etwa für den Fall, dass die Replikation fehlerhaft verlaufen ist (s. oben). Die Aktivität der Kinasen schwankt mit dem Zellzyklus. So ist am G2/M-Übergang
die cdk-Aktivität hoch, und Proteine, welche die
Vorgänge der frühen Mitose steuern, werden vermehrt aktiviert (Abbau der Kernhülle, Kondensation
der Chromosomen, Aufbau der Spindeltubuli).  Als Mitose bezeichnet man die Aufteilung des Chromosomensatzes auf zwei Tochterkerne, als Zytokinese die Aufteilung des Zytoplasmas auf zwei Tochterzellen.
Als Mitose bezeichnet man die Aufteilung des Chromosomensatzes auf zwei Tochterkerne, als Zytokinese die Aufteilung des Zytoplasmas auf zwei Tochterzellen. Differenzierung: Manche (ausdifferenzierte) Zellen, wie Nerven- oder Muskelzellen, teilen sich im Körper gar nicht mehr; sie verlängern ihre G1-Phase - die dann als G0-Phase bezeichnet wird - auf unbestimmte Zeit. Eine Rückkehr in die G1-Phase
ist bei Bedarf möglich (z.B. Lymphozyten, Hepatozyten); wenn sich die
Zelle endgültig differenziert, gibt es aber kein Zurück zum Teilungszyklus
mehr. Wie sich die Zelle entscheidet, hängt von ihrer Umgebung ab (Anwesenheit von Adhäsionsmolekülen / Wachstumsfaktoren).
Differenzierung: Manche (ausdifferenzierte) Zellen, wie Nerven- oder Muskelzellen, teilen sich im Körper gar nicht mehr; sie verlängern ihre G1-Phase - die dann als G0-Phase bezeichnet wird - auf unbestimmte Zeit. Eine Rückkehr in die G1-Phase
ist bei Bedarf möglich (z.B. Lymphozyten, Hepatozyten); wenn sich die
Zelle endgültig differenziert, gibt es aber kein Zurück zum Teilungszyklus
mehr. Wie sich die Zelle entscheidet, hängt von ihrer Umgebung ab (Anwesenheit von Adhäsionsmolekülen / Wachstumsfaktoren).  Demethylierung der DNA und Acetylierung von Histonen bewirken eine "offene" Konformation mit Ablesbarkeit betreffender Genorte,
Demethylierung der DNA und Acetylierung von Histonen bewirken eine "offene" Konformation mit Ablesbarkeit betreffender Genorte,  Methylierung
der DNA und Deacetylierung von Histonen hingegen eine "geschlossene"
Konformation mit behinderter Transkription (Gene "abgeschaltet").
Methylierung
der DNA und Deacetylierung von Histonen hingegen eine "geschlossene"
Konformation mit behinderter Transkription (Gene "abgeschaltet"). Ein Mitogen
ist eine Substanz - meist ein Protein -, die Zellen zur Teilung anregt.
Dabei werden Wege zur Signaltransduktion stimuliert, in welche mitogenaktivierte Proteinkinasen (MAPK, MAP-Kinasen)
involviert sind. Diese aktivieren die Ablesung von Genen, u.a. für den
Transkriptionsfaktor MYC (ein zentraler Expressionsregulator), der
wiederum zahlreiche weitere Gene - z.B. für Cyclin A - aktiviert, was
schließlich das Fortschreiten der S-Phase vorantreibt.
Ein Mitogen
ist eine Substanz - meist ein Protein -, die Zellen zur Teilung anregt.
Dabei werden Wege zur Signaltransduktion stimuliert, in welche mitogenaktivierte Proteinkinasen (MAPK, MAP-Kinasen)
involviert sind. Diese aktivieren die Ablesung von Genen, u.a. für den
Transkriptionsfaktor MYC (ein zentraler Expressionsregulator), der
wiederum zahlreiche weitere Gene - z.B. für Cyclin A - aktiviert, was
schließlich das Fortschreiten der S-Phase vorantreibt. MAP-Kinasen
regulieren neben der Mitosetätigkeit auch Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose der Zelle.
MAP-Kinasen
regulieren neben der Mitosetätigkeit auch Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose der Zelle. Mitogene sind extrazelluläre Signalstoffe, die typischerweise
intrazelluläre "Bremser" des ersten Kontrollpunktes (G1) "überstimmen"
und dadurch den Eingang zur Synthesephase der betreffenden Zelle
öffnen. Sie tun dies, indem sie an Tyrosinkinase-Rezeptoren in der
Zellmembran binden und so intrazelluläre Folgeraktionen (über Ras, MAP)
anstoßen, die in der Freigabe der Mitose münden.
Mitogene sind extrazelluläre Signalstoffe, die typischerweise
intrazelluläre "Bremser" des ersten Kontrollpunktes (G1) "überstimmen"
und dadurch den Eingang zur Synthesephase der betreffenden Zelle
öffnen. Sie tun dies, indem sie an Tyrosinkinase-Rezeptoren in der
Zellmembran binden und so intrazelluläre Folgeraktionen (über Ras, MAP)
anstoßen, die in der Freigabe der Mitose münden.
 Abbildung: Mitosezyklus
Abbildung: Mitosezyklus
 Bei der Zellteilung entfernen sich Zentriolen voneinander (bedingt durch Mikrotubuli,
die zwischen ihnen entstehen) und beteiligen sich am Aufbau eines
Spindelapparates, der jeweils zwei Chromatidensätze voneinander trennt.
Dabei greifen die Mikrotubuli des Spindelapparates über als Kinetochor
Bei der Zellteilung entfernen sich Zentriolen voneinander (bedingt durch Mikrotubuli,
die zwischen ihnen entstehen) und beteiligen sich am Aufbau eines
Spindelapparates, der jeweils zwei Chromatidensätze voneinander trennt.
Dabei greifen die Mikrotubuli des Spindelapparates über als Kinetochor bezeichnete Proteinkomplexe an Zentromeren
bezeichnete Proteinkomplexe an Zentromeren an, das sind bis zu 106 Nukleotidpaare lange, stark repetitive DNA-Sequenzen der Chromosomen, an denen diese Verankerung stattfinden kann.
an, das sind bis zu 106 Nukleotidpaare lange, stark repetitive DNA-Sequenzen der Chromosomen, an denen diese Verankerung stattfinden kann.  Das Zentrosom ist ist eine Zellorganelle, welche Mikrotubuli organisiert. Es besteht aus zwei rechtwinklig zueinander angeordneten Zentriolen, umgeben von einem hochstrukturierten Proteinkomplex (microtubule-organizing center MTOC), das den Spindelapparat bzw. Zilien ordnet. Zentromeren sind Chromosomenregionen, die während einer Zellteilung zwei homologe Chromosomen miteinander verbinden. An diesen bilden sich Kinetochoren aus,
das sind Proteinkomplexe, an denen - während Zellteilungen (Mitosen,
Meiosen) - Mikrotubuli der Mitosespindel befestigt sind.
Das Zentrosom ist ist eine Zellorganelle, welche Mikrotubuli organisiert. Es besteht aus zwei rechtwinklig zueinander angeordneten Zentriolen, umgeben von einem hochstrukturierten Proteinkomplex (microtubule-organizing center MTOC), das den Spindelapparat bzw. Zilien ordnet. Zentromeren sind Chromosomenregionen, die während einer Zellteilung zwei homologe Chromosomen miteinander verbinden. An diesen bilden sich Kinetochoren aus,
das sind Proteinkomplexe, an denen - während Zellteilungen (Mitosen,
Meiosen) - Mikrotubuli der Mitosespindel befestigt sind.  Abbildung):
Abbildung): Prophase:
Während der Prophase replizieren die Chromosomen und werden zu zwei
Chromatiden, die über ein Kinetochor miteinander verknüpft sind. Die
DNA kondensiert zu kompakten, stabförmigen
Schwesterchromatiden, die zunächst verbunden bleiben (Kohäsion). Der
Spindelapparat beginnt sich zu formieren, und Zentrosomen wandern an
entgegengesetzte Zellpole. Diese Phase kann zwischen
einer halben und (seltener) mehreren Stunden dauern.
Prophase:
Während der Prophase replizieren die Chromosomen und werden zu zwei
Chromatiden, die über ein Kinetochor miteinander verknüpft sind. Die
DNA kondensiert zu kompakten, stabförmigen
Schwesterchromatiden, die zunächst verbunden bleiben (Kohäsion). Der
Spindelapparat beginnt sich zu formieren, und Zentrosomen wandern an
entgegengesetzte Zellpole. Diese Phase kann zwischen
einer halben und (seltener) mehreren Stunden dauern. Prometaphase:
Der Zellkern verliert seine Hülle, die mitotische Spindel haftet sich
an die Kinetochoren (in der Abbildung als gelbe Punkte dargestellt) der
Chromosomen. Die Spindel - aufgebaut aus Mikrotubuli (bestehend aus α- und β-Tubulin) - kann nicht direkt an ein Zentromer binden, sondern benötigt das Kinetochor als "Vermittler".
Prometaphase:
Der Zellkern verliert seine Hülle, die mitotische Spindel haftet sich
an die Kinetochoren (in der Abbildung als gelbe Punkte dargestellt) der
Chromosomen. Die Spindel - aufgebaut aus Mikrotubuli (bestehend aus α- und β-Tubulin) - kann nicht direkt an ein Zentromer binden, sondern benötigt das Kinetochor als "Vermittler". Metaphase:
Der an den Zentriolen entspringende Spindelapparat entwickelt sich, und
die Schwesterchromatiden heften sich paarweise an die Mitosespindel und
werden in der Äquatorialebene angeordnet. In diesem Zeitabschnitt sind
die Chromosomen am stärksten kondensiert (lichtmikroskopisch gut sichtbar). Die Metaphase
dauert etwa 10 Minuten.
Metaphase:
Der an den Zentriolen entspringende Spindelapparat entwickelt sich, und
die Schwesterchromatiden heften sich paarweise an die Mitosespindel und
werden in der Äquatorialebene angeordnet. In diesem Zeitabschnitt sind
die Chromosomen am stärksten kondensiert (lichtmikroskopisch gut sichtbar). Die Metaphase
dauert etwa 10 Minuten. Anaphase:
In dieser 2 bis 20 Minuten dauernden Phase verschwindet die Schwesterchromatiden-Kohäsion, die Chromatiden
trennen sich mit Hilfe der Spindelfasern und werden zu den Spindelpolen gezogen. In der Mitte der Zelle bildet sich ein
gürtelförmiger kontraktiler Ring aus Aktinfilamenten, der die Trennung in zwei Zellen unterstützt (Zytokinese). Die Kohäsion der Chromatiden wird in der Anaphase durch anaphase-promoting complexes
(APCs) aufgelöst und die Trennung der Chromatiden ermöglicht. Ein APC
besteht aus mehreren Untereinheiten, die z.T. Ubiquitinaktivität haben
und so u.a. Cycline abbaut.
Anaphase:
In dieser 2 bis 20 Minuten dauernden Phase verschwindet die Schwesterchromatiden-Kohäsion, die Chromatiden
trennen sich mit Hilfe der Spindelfasern und werden zu den Spindelpolen gezogen. In der Mitte der Zelle bildet sich ein
gürtelförmiger kontraktiler Ring aus Aktinfilamenten, der die Trennung in zwei Zellen unterstützt (Zytokinese). Die Kohäsion der Chromatiden wird in der Anaphase durch anaphase-promoting complexes
(APCs) aufgelöst und die Trennung der Chromatiden ermöglicht. Ein APC
besteht aus mehreren Untereinheiten, die z.T. Ubiquitinaktivität haben
und so u.a. Cycline abbaut. Telophase:
Die Spindel zerfällt, Kernhülle und neue Nukleoli werden
wiederhergestellt, die
Chromosomen in neu gebildete Zellkerne eingefügt und dekondensiert
(entspiralisiert), womit sie wieder in den "Arbeitszustand"
treten können (Ablesung der Erbinformation, die während der Mitose sehr
eingeschränkt ist). Mit der Wiederherstellung der Kernmembran ist die
Telophase abgeschlossen.
Telophase:
Die Spindel zerfällt, Kernhülle und neue Nukleoli werden
wiederhergestellt, die
Chromosomen in neu gebildete Zellkerne eingefügt und dekondensiert
(entspiralisiert), womit sie wieder in den "Arbeitszustand"
treten können (Ablesung der Erbinformation, die während der Mitose sehr
eingeschränkt ist). Mit der Wiederherstellung der Kernmembran ist die
Telophase abgeschlossen.
 Die Interphase besteht aus
Die Interphase besteht aus Während der M-Phase (mitosis) teilt sich die Zelle
Während der M-Phase (mitosis) teilt sich die Zelle In der G0-Phase hat die Zelle den Teilungszyklus verlassen ("Ruhepause" / endgültige Differenzierung / Alterung)
In der G0-Phase hat die Zelle den Teilungszyklus verlassen ("Ruhepause" / endgültige Differenzierung / Alterung) Das Genom ist die vollständige DNA-Sequenz eines Organismus (bei Viren auch RNA). Das Genom des Menschen ist erstaunlich klein: Es entspricht etwa 750 Megabyte (3.109 DNA-Nukleotide mit jeweils 4 möglichen Basen pro Nukleotid). Die Gesamtzahl der Gene (inklusive Pseudogene) liegt bei ~54.103 (zum Vergleich: E. coli ~4,5.103,
Taufliege ~13,5.103, Wasserfloh ~31.103, Gemüsekohl ~100.103 -
Genomgröße
und Komplexität des Organismus sind kaum korreliert).
Das Genom ist die vollständige DNA-Sequenz eines Organismus (bei Viren auch RNA). Das Genom des Menschen ist erstaunlich klein: Es entspricht etwa 750 Megabyte (3.109 DNA-Nukleotide mit jeweils 4 möglichen Basen pro Nukleotid). Die Gesamtzahl der Gene (inklusive Pseudogene) liegt bei ~54.103 (zum Vergleich: E. coli ~4,5.103,
Taufliege ~13,5.103, Wasserfloh ~31.103, Gemüsekohl ~100.103 -
Genomgröße
und Komplexität des Organismus sind kaum korreliert).  ) oder kompakt, dicht gepackt und schwer bis gar nicht ablesbar (silent state, Heterochromatin
) oder kompakt, dicht gepackt und schwer bis gar nicht ablesbar (silent state, Heterochromatin  ).
). 
 Abbildung: Heterochromatin und Euchromatin
Abbildung: Heterochromatin und Euchromatin Heterochromatin
beinhaltet im "Arbeitszustand" der Zelle (Interphasenkern) etwa 10% des gesamten Genoms und stellt unterschiedliche Formen der
Chromatinverdichtung dar, die sich auch unterschiedlich auf die
Expression von Genen auswirken - im allgemeinen inaktivierend. Konstitutives Heterochromatin ist immer dicht gepackt (vor allem im Bereich der Zentrosomen und Telomere) und enthält wenige Gene, fakultatives Heterochromatin kann dekondensiert werden und ist reich an Genen. Heterochromatin weist stark gebundenes H1-Histon auf, das den DNA-Faden an Nukleosom fixiert.
Heterochromatin
beinhaltet im "Arbeitszustand" der Zelle (Interphasenkern) etwa 10% des gesamten Genoms und stellt unterschiedliche Formen der
Chromatinverdichtung dar, die sich auch unterschiedlich auf die
Expression von Genen auswirken - im allgemeinen inaktivierend. Konstitutives Heterochromatin ist immer dicht gepackt (vor allem im Bereich der Zentrosomen und Telomere) und enthält wenige Gene, fakultatives Heterochromatin kann dekondensiert werden und ist reich an Genen. Heterochromatin weist stark gebundenes H1-Histon auf, das den DNA-Faden an Nukleosom fixiert.  Euchromatin
hingegen bindet H1-Histon relativ schwach und zeigt stark methylierte
Histone der Nukleosomenkerne, was mit guter Ablesbarkeit der DNA
korreliert. In differenzierten Zellen ist das Euchromatin nicht
durchgehend "gelockert", sondern unterschiedlich stark kondensiert. Das
Muster der Ablesbarkeit von Genen bestimmt, welche Proteine
synthetisiert werden können und spiegelt die Differenzierung der Zelle
(Myozyt? Lymphozyt? Hepatozyt? etc) wider.
Euchromatin
hingegen bindet H1-Histon relativ schwach und zeigt stark methylierte
Histone der Nukleosomenkerne, was mit guter Ablesbarkeit der DNA
korreliert. In differenzierten Zellen ist das Euchromatin nicht
durchgehend "gelockert", sondern unterschiedlich stark kondensiert. Das
Muster der Ablesbarkeit von Genen bestimmt, welche Proteine
synthetisiert werden können und spiegelt die Differenzierung der Zelle
(Myozyt? Lymphozyt? Hepatozyt? etc) wider.
 Abbildung: Epigenetische Veränderung - Beispiel Histonmodifikation
Abbildung: Epigenetische Veränderung - Beispiel Histonmodifikation Das Epigenom bezieht sich auf
Veränderungen an DNA und Histonproteinen, die nicht die Basensequenz
betreffen und u.a. durch DNA-Methylierung, modifizierte Ablesbarkeit
oder Einfluss auf die Genexpression erklärbar sind.
Das Epigenom bezieht sich auf
Veränderungen an DNA und Histonproteinen, die nicht die Basensequenz
betreffen und u.a. durch DNA-Methylierung, modifizierte Ablesbarkeit
oder Einfluss auf die Genexpression erklärbar sind.

 Abbildung: Grad der DNA-Methylierung als Funktion der individuellen Entwicklung
Abbildung: Grad der DNA-Methylierung als Funktion der individuellen Entwicklung
 die DNA durch Basen-Methylierung (DNA-Methyltransferasen) - die Base bleibt
erhalten, es handelt sich also um keine Mutation. Die Modifikationen sind reversibel (Demethylierung durch DNA-Demethylasen etc), können aber über längere Zeit erhalten bleiben und bilden so die Basis für ein "Zellgedächtnis". Die Dynamik der Methylierungen ist vermutlich der wichtigste epigenetische
Mechanismus
die DNA durch Basen-Methylierung (DNA-Methyltransferasen) - die Base bleibt
erhalten, es handelt sich also um keine Mutation. Die Modifikationen sind reversibel (Demethylierung durch DNA-Demethylasen etc), können aber über längere Zeit erhalten bleiben und bilden so die Basis für ein "Zellgedächtnis". Die Dynamik der Methylierungen ist vermutlich der wichtigste epigenetische
Mechanismus Histon-Endstücke (
Histon-Endstücke ( Abbildung oben) durch Anfügen (Writer - z.B. Acetylierung,
Methylierung oder Phosphorylierung), Entfernen (Eraser, z.B. Demethylierung durch Histon-Demethylasen) oder
"Verwalten" von Modifikationen. Modifikationen des Histonmoleküls
können Genaktivitäten verändern und Proteinkomplexe (Reader, Leser-Proteine)
attrahieren. Haben solche Leserkomplexe an den modifizierten
Histonkomplex angedockt, ziehen sie weitere Proteinkomplexe an,
die enzymatisch aktiv sind oder zusätzliche Bindungsstellen
bereitstellen und so die Aktivität abhängiger Gene verändern
Abbildung oben) durch Anfügen (Writer - z.B. Acetylierung,
Methylierung oder Phosphorylierung), Entfernen (Eraser, z.B. Demethylierung durch Histon-Demethylasen) oder
"Verwalten" von Modifikationen. Modifikationen des Histonmoleküls
können Genaktivitäten verändern und Proteinkomplexe (Reader, Leser-Proteine)
attrahieren. Haben solche Leserkomplexe an den modifizierten
Histonkomplex angedockt, ziehen sie weitere Proteinkomplexe an,
die enzymatisch aktiv sind oder zusätzliche Bindungsstellen
bereitstellen und so die Aktivität abhängiger Gene verändern die Zugänglichkeit an die DNA durch strukturelle Veränderungen des Chromatins
die Zugänglichkeit an die DNA durch strukturelle Veränderungen des Chromatins die Genexpression, beeinflusst durch Interaktion von Histonen und Proteinen. Genexpression nennt man die Transkription eines Gens zu mRNA, seine Translation zu Protein, oder beides.
die Genexpression, beeinflusst durch Interaktion von Histonen und Proteinen. Genexpression nennt man die Transkription eines Gens zu mRNA, seine Translation zu Protein, oder beides.
 Abbildung: Zellzyklus (vereinfacht)
Abbildung: Zellzyklus (vereinfacht)
 Abbildung), man spricht von homologen Chromosomen (Homologen). Weiters hat die Frau zwei
X-Chromosomen, der Mann ein X- und ein Y-Chromosom
(Heterosomen,
“Geschlechtschromosomen”). Beim Mann stammt das X-Chromosom von der
Mutter und das Y-Chromosom vom Vater (nichthomologes Chromosomenpaar).
Abbildung), man spricht von homologen Chromosomen (Homologen). Weiters hat die Frau zwei
X-Chromosomen, der Mann ein X- und ein Y-Chromosom
(Heterosomen,
“Geschlechtschromosomen”). Beim Mann stammt das X-Chromosom von der
Mutter und das Y-Chromosom vom Vater (nichthomologes Chromosomenpaar). Über Meiose s. dort
Über Meiose s. dort Die
Frage, wie Proteine in der Zelle für zytosolische oder sekretorische
Verwendung "sortiert" werden, wurde u.a. vom US-amerikanischen
Biochemiker Günter Blobel
aufgeklärt, der dafür 1999 den Nobelpreis für Physiologie oder Medizin
erhielt. Die Sortierung hängt davon ab, ob die mRNA mit einer Signalsequenz (für das endoplasmatische Retikulum) ausgestattet ist oder nicht. Das "Protein Targeting" erfolgt dann über Translokationssequenzen
- diese bestimmen, wohin (in welches zelluläre Kompartiment) das
gebildete Protein genau gelangt (z.B. in den Zellkern, die
Mitochondrien, etc).
Die
Frage, wie Proteine in der Zelle für zytosolische oder sekretorische
Verwendung "sortiert" werden, wurde u.a. vom US-amerikanischen
Biochemiker Günter Blobel
aufgeklärt, der dafür 1999 den Nobelpreis für Physiologie oder Medizin
erhielt. Die Sortierung hängt davon ab, ob die mRNA mit einer Signalsequenz (für das endoplasmatische Retikulum) ausgestattet ist oder nicht. Das "Protein Targeting" erfolgt dann über Translokationssequenzen
- diese bestimmen, wohin (in welches zelluläre Kompartiment) das
gebildete Protein genau gelangt (z.B. in den Zellkern, die
Mitochondrien, etc). vgl. dort
vgl. dort Abbildung):
Abbildung):
 Abbildung: Aufbau und Funktion des Zellkerns
Abbildung: Aufbau und Funktion des Zellkerns
 Aufbau und Arten der RNA
Aufbau und Arten der RNA  Translation
Translation  Transfer in raues EPR
Transfer in raues EPR Als Transkription bezeichnet man das Kopieren von Genen durch Bildung entsprechender RNA. Nicht jede Transkription führt auch zu Translation, also der Synthese entsprechender Proteine. Transkription bedeutet, dass entsprechende Gene exprimiert werden (Genexpression). Unter einem Transkriptom versteht man die Gesamtheit aller zu einem bestimmten Zeitpunkt von einer Zelle hergestellten RNS-Moleküle.
Als Transkription bezeichnet man das Kopieren von Genen durch Bildung entsprechender RNA. Nicht jede Transkription führt auch zu Translation, also der Synthese entsprechender Proteine. Transkription bedeutet, dass entsprechende Gene exprimiert werden (Genexpression). Unter einem Transkriptom versteht man die Gesamtheit aller zu einem bestimmten Zeitpunkt von einer Zelle hergestellten RNS-Moleküle.  Abbildung: Transkription (Bildung von mRNA) und Translation (Proteinsynthese)
Abbildung: Transkription (Bildung von mRNA) und Translation (Proteinsynthese)

 Abbildung: Modulation der Transkription
Abbildung: Modulation der Transkription
 Abbildung). Dadurch
verändern sich Chromatinstruktur, Aktivität der RNA-Polymerase und
Bindung von Transkriptionsfaktoren. Resultat ist die Verstärkung (oder
Abschwächung) der Proteinsynthese.
Abbildung). Dadurch
verändern sich Chromatinstruktur, Aktivität der RNA-Polymerase und
Bindung von Transkriptionsfaktoren. Resultat ist die Verstärkung (oder
Abschwächung) der Proteinsynthese.
 Abbildung: Multiple Genregulation
Abbildung: Multiple Genregulation
 Abbildung).
Abbildung).  Codierende RNA, das sind Boten-RNA (messenger RNA, mRNA), das Produkt der Transkription
(Ablesung der DNA im Zellkern oder in Mitochondrien). mRNA dienen als
Informationsträger für die Synthese von Proteinen (Translation im
Zytoplasma oder in Mitochondrien) und sind (je nach zu codierendem Protein) unterschiedlich lang (bis zu mehrere tausend Basenpaare).
Codierende RNA, das sind Boten-RNA (messenger RNA, mRNA), das Produkt der Transkription
(Ablesung der DNA im Zellkern oder in Mitochondrien). mRNA dienen als
Informationsträger für die Synthese von Proteinen (Translation im
Zytoplasma oder in Mitochondrien) und sind (je nach zu codierendem Protein) unterschiedlich lang (bis zu mehrere tausend Basenpaare).  Nicht-codierende RNA, eine heterogene Gruppe von Ribonukleinsäuren, die nicht als Bauanleitung für Proteine dienen. Nichtcodierende DNA sind u.a.
Nicht-codierende RNA, eine heterogene Gruppe von Ribonukleinsäuren, die nicht als Bauanleitung für Proteine dienen. Nichtcodierende DNA sind u.a. Ribosomale (rRNA) als Bestandteile von
Ribosomen
Ribosomale (rRNA) als Bestandteile von
Ribosomen Transfer-RNA (tRNA), welche die jeweils erforderlichen Aminosäuren an den
Transkriptionsvorgang anliefern
Transfer-RNA (tRNA), welche die jeweils erforderlichen Aminosäuren an den
Transkriptionsvorgang anliefern Kleine (kurze) RNAs (sRNA, small RNA) zur Regulierung spezifischer Zielgene, zum Spleißen von RNA u.a.
Kleine (kurze) RNAs (sRNA, small RNA) zur Regulierung spezifischer Zielgene, zum Spleißen von RNA u.a. Mikro-RNAs (miRNA, microRNA) beteiligen sich an der Regulierung von Transkription und Translation
Mikro-RNAs (miRNA, microRNA) beteiligen sich an der Regulierung von Transkription und Translation  Nichtkodierende lange RNA (lncRNA, long non-coding RNA)
- definiert als nichtcodierende RNA mit über 200 Nukleotiden mit
Funktionen in der Regulierung von Transkription und
posttranskriptionalen Vorgängen.
Nichtkodierende lange RNA (lncRNA, long non-coding RNA)
- definiert als nichtcodierende RNA mit über 200 Nukleotiden mit
Funktionen in der Regulierung von Transkription und
posttranskriptionalen Vorgängen.

 Abbildung:
Struktur einer Boten- (m) RNA
Abbildung:
Struktur einer Boten- (m) RNA

 Anbringen
eines methylierten Guanosinmoleküls (5'-Kappe) am 5'-Ende (hilft wahrscheinlich beim Spleißen, beim Transport aus dem Zellkern und beim "Einfädeln" der Translation im Zytoplasma),
Anbringen
eines methylierten Guanosinmoleküls (5'-Kappe) am 5'-Ende (hilft wahrscheinlich beim Spleißen, beim Transport aus dem Zellkern und beim "Einfädeln" der Translation im Zytoplasma), Anbringen eines Poly-A-Schwanzes am 3'-Ende (Funktion: RNA-Stabilisierung, Hilfe beim Transport durch den Zellkern),
Anbringen eines Poly-A-Schwanzes am 3'-Ende (Funktion: RNA-Stabilisierung, Hilfe beim Transport durch den Zellkern), Abbildung):
Abbildung):  Dem 5'-Pol mit einer - vor dem Abbau durch Nukleasen schützenden - "Kappe" (Cap),
Dem 5'-Pol mit einer - vor dem Abbau durch Nukleasen schützenden - "Kappe" (Cap),  einer darauf folgenden nicht proteincodierenden Sequenz (5'-UTR: untranslated region) - diese beeinflusst u.a. Stabilität, Export und Effizienz der mRNA,
einer darauf folgenden nicht proteincodierenden Sequenz (5'-UTR: untranslated region) - diese beeinflusst u.a. Stabilität, Export und Effizienz der mRNA,  dem peptid- bzw. proteincodierenden Leseraster (ORF: Open reading frame) - dieser beginnt mit einem Start-Codon (AUG) und endet mit einem
Stop-Codon (UAA, UAG oder UGA). Die Translation (Proteinsynthese)
beginnt mit dem Start-Codon und endet nach dem Stop-Codon,
dem peptid- bzw. proteincodierenden Leseraster (ORF: Open reading frame) - dieser beginnt mit einem Start-Codon (AUG) und endet mit einem
Stop-Codon (UAA, UAG oder UGA). Die Translation (Proteinsynthese)
beginnt mit dem Start-Codon und endet nach dem Stop-Codon, einer nicht proteincodierenden 3'-UTR (three prime untranslated region), welche direkt dem Stop-Codon folgt und auf mehrfache Art und Weise die Genexpression oder die Stabilität des Transkriptionsprodukts beeinflusst, sowie
einer nicht proteincodierenden 3'-UTR (three prime untranslated region), welche direkt dem Stop-Codon folgt und auf mehrfache Art und Weise die Genexpression oder die Stabilität des Transkriptionsprodukts beeinflusst, sowie  einem durch Polyadenylierung entstandenen Poly-A-Schwanz (Poly (A) tail) am 5'-Ende der mRNA.
einem durch Polyadenylierung entstandenen Poly-A-Schwanz (Poly (A) tail) am 5'-Ende der mRNA. s. oben):
s. oben): Mikro-RNA (miRNA, µRNA)
- aus 21-23 Nukleotiden bestehende RNA-Fragmente, welche die Expression
ihrer Zielgene stoppen können (Gen-Stilllegung durch RNA-Interferenz,
ein phylogenetisch sehr alter Mechanismus). Sie setzen sich auf
komplementäre DNS-Sequenzen einer mRNA und lassen sie auf verschiedene Art und Weise "verstummen" (gene silencing), indem sie z.B. die Translation behindern (beim Menschen ist die mRNA-Destabilisierung der primäre Wirkungsmechanismus):
Mikro-RNA (miRNA, µRNA)
- aus 21-23 Nukleotiden bestehende RNA-Fragmente, welche die Expression
ihrer Zielgene stoppen können (Gen-Stilllegung durch RNA-Interferenz,
ein phylogenetisch sehr alter Mechanismus). Sie setzen sich auf
komplementäre DNS-Sequenzen einer mRNA und lassen sie auf verschiedene Art und Weise "verstummen" (gene silencing), indem sie z.B. die Translation behindern (beim Menschen ist die mRNA-Destabilisierung der primäre Wirkungsmechanismus):
 Abbildung: Steuerung der Genexpression durch miRNA
Abbildung: Steuerung der Genexpression durch miRNA
 Abbildung
geschilderten Weise eine wichtige Rolle für die Steuerung des
Zellstoffwechsels spielen und Veränderungen ihrer Funktion diverse
Krankheitsbilder bedingen.
Abbildung
geschilderten Weise eine wichtige Rolle für die Steuerung des
Zellstoffwechsels spielen und Veränderungen ihrer Funktion diverse
Krankheitsbilder bedingen. Kleine Kern- (small nuclear) RNA (snRNA)
sind am Spleißen von prä-mRNA zu mRNA beteiligt. Sie bestehen aus 20-25
Nikleotiden, die ebenfalls mit der Genexpression interferieren. Soll die Expression eines Proteins (dessen genetische DNA-Basensequenz bekannt ist) beeinflusst werden, wäre dies mittels entsprechend komplementärer snRNA möglich.
Kleine Kern- (small nuclear) RNA (snRNA)
sind am Spleißen von prä-mRNA zu mRNA beteiligt. Sie bestehen aus 20-25
Nikleotiden, die ebenfalls mit der Genexpression interferieren. Soll die Expression eines Proteins (dessen genetische DNA-Basensequenz bekannt ist) beeinflusst werden, wäre dies mittels entsprechend komplementärer snRNA möglich. Kleine Interferenz-RNA (siRNA, small interfering RNA), auch silencing RNA, 20-25 Basenpaare lang, funktionieren ähnlich wie Mikro-RNA - sie
können die Translation von Zielgenen blockieren. Auch sie nutzen den
Dicer-Mechanismus, können aber auch die Genexpression fördern.
Kleine Interferenz-RNA (siRNA, small interfering RNA), auch silencing RNA, 20-25 Basenpaare lang, funktionieren ähnlich wie Mikro-RNA - sie
können die Translation von Zielgenen blockieren. Auch sie nutzen den
Dicer-Mechanismus, können aber auch die Genexpression fördern. Zahlreiche weitere sRNA sind bekannt, z.B. Piwi-interacting RNA (piRNA - Abkürzung für P-element induced wimpy testis) - beim Menschen notwendig für die Spermatogenese; kleine nukleoläre RNA (snoRNA, small nucleolar RNA) - machen Ribosomen fit für die Translation und snRNAs für das Splicing; etc.
Zahlreiche weitere sRNA sind bekannt, z.B. Piwi-interacting RNA (piRNA - Abkürzung für P-element induced wimpy testis) - beim Menschen notwendig für die Spermatogenese; kleine nukleoläre RNA (snoRNA, small nucleolar RNA) - machen Ribosomen fit für die Translation und snRNAs für das Splicing; etc.
 Abbildung: Schema der Eiweißsynthese
Abbildung: Schema der Eiweißsynthese
 Der zytoplasmatische Weg (cytoplasmic pathway) dient
der Synthese von Eiweiß für Zellkern, Zytoplasma, Mitochondrien und
Peroxisomen. Spezifische Translokationssignale fördern die Proteinbildung für Zellkern, Mitochondrien oder Peroxisomen - oder ein
Translokationssignal fehlt überhaupt, und das Protein wird direkt in das Zytoplasma
abgegeben, wo die gesamte Translation (Proteinsynthese) abläuft.
Der zytoplasmatische Weg (cytoplasmic pathway) dient
der Synthese von Eiweiß für Zellkern, Zytoplasma, Mitochondrien und
Peroxisomen. Spezifische Translokationssignale fördern die Proteinbildung für Zellkern, Mitochondrien oder Peroxisomen - oder ein
Translokationssignal fehlt überhaupt, und das Protein wird direkt in das Zytoplasma
abgegeben, wo die gesamte Translation (Proteinsynthese) abläuft.  Der sekretorische Weg (secretory pathway)
dient der Synthese von Sekreten oder von integralen Membranproteinen, Eiweiß für das endoplasmatische Retikulum oder für
Lysosomen. Für diesen Weg
rücken Ribosomen an die Wand des rauen endoplasmatischen Retikulums:
Der sekretorische Weg (secretory pathway)
dient der Synthese von Sekreten oder von integralen Membranproteinen, Eiweiß für das endoplasmatische Retikulum oder für
Lysosomen. Für diesen Weg
rücken Ribosomen an die Wand des rauen endoplasmatischen Retikulums: Animation).
Animation).

 Animation: Ablauf der Synthese eines für den Export bestimmten Proteins
Animation: Ablauf der Synthese eines für den Export bestimmten Proteins
 Abbildung dargestellten Mechanismus schrittweise in und durch die
Membran hindurch. Die Kanäle (Translocons),
durch welche die aus den Ribosomen wachsenden - hydrophilen -
Aminosäureketten "gesteckt" (transloziert) werden, ersparen diesen
einen direkten Kontakt mit hydrophobem Membranmaterial.
Abbildung dargestellten Mechanismus schrittweise in und durch die
Membran hindurch. Die Kanäle (Translocons),
durch welche die aus den Ribosomen wachsenden - hydrophilen -
Aminosäureketten "gesteckt" (transloziert) werden, ersparen diesen
einen direkten Kontakt mit hydrophobem Membranmaterial. Abbildung
- stellt sicher, dass die Synthese von Membran- und sekretorische
Proteine nur dann fortgesetzt wird, wenn für sie ein
Translokationskanal (ein Translocon) frei ist (cotranslational translocation).
Abbildung
- stellt sicher, dass die Synthese von Membran- und sekretorische
Proteine nur dann fortgesetzt wird, wenn für sie ein
Translokationskanal (ein Translocon) frei ist (cotranslational translocation). 
 Abbildung: Herstellung eines sekretorischen Proteins (vgl. <Animation)
Abbildung: Herstellung eines sekretorischen Proteins (vgl. <Animation)
 Abbildung). Dann hat das Protein seine endgültige Form erhalten
und kann exozytiert werden (ein Vorgang, der durch Calciumionen
angeregt wird).
Abbildung). Dann hat das Protein seine endgültige Form erhalten
und kann exozytiert werden (ein Vorgang, der durch Calciumionen
angeregt wird). Um
Proteine in Vesikel zu bringen (von wo sie exozytiert werden können),
bedarf es der Beteiligung des rauen endoplasmatischen Retikulums
(Proteinsynthese) und des Golgi-Apparats (Verbringung in Vesikel).
Um
Proteine in Vesikel zu bringen (von wo sie exozytiert werden können),
bedarf es der Beteiligung des rauen endoplasmatischen Retikulums
(Proteinsynthese) und des Golgi-Apparats (Verbringung in Vesikel).
 Abbildung: Synthese, Sortierung und Freisetzung von Peptiden
Abbildung: Synthese, Sortierung und Freisetzung von Peptiden
 Wie
können sich Zellen auf Veränderungen der Umwelt anpassen? Ein Mechanismus besteht darin, die Transkription und Translation
zu steuern und/oder deren molekularen Ergebnisse (Ribonukleinsäure,
Protein) zu modifizieren. Nur ein kleiner Teil der vorhandenen Gene
wird auch exprimiert. Die Exprimierungsmuster können wechseln (s. oben unter "Genexpression").
Wie
können sich Zellen auf Veränderungen der Umwelt anpassen? Ein Mechanismus besteht darin, die Transkription und Translation
zu steuern und/oder deren molekularen Ergebnisse (Ribonukleinsäure,
Protein) zu modifizieren. Nur ein kleiner Teil der vorhandenen Gene
wird auch exprimiert. Die Exprimierungsmuster können wechseln (s. oben unter "Genexpression").  s. dort).
s. dort). 
 Als Rb-Protein (pRB,
Retinoblastom-Protein) bezeichnet man ein physiologisches Protein, das
in den Zellzyklus regulierend eingreift, indem es die Aktivität von Transkriptionsfaktoren unterdrückt: Es verhindert u.a. übertriebenes Zellwachstum, indem es den Teilungszyklus
aufhält, bis die Zelle bereit für eine Mitose ist. Auf diese Weise kann
es auch die Bildung von Tumoren verhindern (der Name stammt daher, dass
Mutationen des Rb-Proteins zur Bildung von Retinoblastomen führen
kann).
Als Rb-Protein (pRB,
Retinoblastom-Protein) bezeichnet man ein physiologisches Protein, das
in den Zellzyklus regulierend eingreift, indem es die Aktivität von Transkriptionsfaktoren unterdrückt: Es verhindert u.a. übertriebenes Zellwachstum, indem es den Teilungszyklus
aufhält, bis die Zelle bereit für eine Mitose ist. Auf diese Weise kann
es auch die Bildung von Tumoren verhindern (der Name stammt daher, dass
Mutationen des Rb-Proteins zur Bildung von Retinoblastomen führen
kann). 
 Abbildung: EGF-Rezeptor-Wirkungen an Zielorganen
Abbildung: EGF-Rezeptor-Wirkungen an Zielorganen
 Abbildung) exprimieren (davon gibt es zahlreiche Spielarten), wachstumsfördernd
und mitosetriggernd (EGF wird für
Zellkulturen verwendet). EGF kommt in zahlreichen Gewebsflüssigkeiten
vor, z.B. auch im Speichelsekret, und hat dort trophische Wirkungen.
EGF spielt für die Wundheilung eine wichtige Rolle: so wird er in
heilenden Hautwunden von Keratinozyten, Makrophagen und anderen
eingewanderten Entzündungszellen produziert.
Abbildung) exprimieren (davon gibt es zahlreiche Spielarten), wachstumsfördernd
und mitosetriggernd (EGF wird für
Zellkulturen verwendet). EGF kommt in zahlreichen Gewebsflüssigkeiten
vor, z.B. auch im Speichelsekret, und hat dort trophische Wirkungen.
EGF spielt für die Wundheilung eine wichtige Rolle: so wird er in
heilenden Hautwunden von Keratinozyten, Makrophagen und anderen
eingewanderten Entzündungszellen produziert. Für die Entdeckung des NGF erhielten die italienische Neurobiologin Rita Levi-Montalcini und der US-Amerikaner Stanley Cohen 1986 den Nobelpreis für Physiologie oder Medizin.
Für die Entdeckung des NGF erhielten die italienische Neurobiologin Rita Levi-Montalcini und der US-Amerikaner Stanley Cohen 1986 den Nobelpreis für Physiologie oder Medizin.
 Abbildung: Wirkungen des PDGF
Abbildung: Wirkungen des PDGF
 Abbildung).
Abbildung). s. auch dort
s. auch dort
 Abbildung: Mechanismen, über welche die extrazelluläre Matrix und
Wachstumsfaktoren zusammenwirken, um Signalkaskaden in der Zelle zu
aktivieren
Abbildung: Mechanismen, über welche die extrazelluläre Matrix und
Wachstumsfaktoren zusammenwirken, um Signalkaskaden in der Zelle zu
aktivieren

 Abbildung: Verletzung und Wundheilung
Abbildung: Verletzung und Wundheilung
 Entzündung (inflammation) - ausgelöst durch Vorgänge der Thrombozytenaktivierung und Gerinnselbildung
Entzündung (inflammation) - ausgelöst durch Vorgänge der Thrombozytenaktivierung und Gerinnselbildung Proliferation
Proliferation
 - das Gerinnsel dient als Matritze für mobile Zellen, die durch
freigesetzte Zytokine und Wachstumsfaktoren angelockt
werden; Fibroblasten wandern ein, Granulationsgewebe
- das Gerinnsel dient als Matritze für mobile Zellen, die durch
freigesetzte Zytokine und Wachstumsfaktoren angelockt
werden; Fibroblasten wandern ein, Granulationsgewebe  formiert sich, das Fibringerinnsel wird durch Fibrinolyse weggeräumt; Neutrophile
und Makrophagen bauen nutzlos gewordene Stoffe ab, entfernen sie,
bekämpfen Bakterien und beteiligen sich an Aufbauprozessen; und es
kommt zu Re-Epithelialisierung
formiert sich, das Fibringerinnsel wird durch Fibrinolyse weggeräumt; Neutrophile
und Makrophagen bauen nutzlos gewordene Stoffe ab, entfernen sie,
bekämpfen Bakterien und beteiligen sich an Aufbauprozessen; und es
kommt zu Re-Epithelialisierung Reifung (maturation) mit Neubildung extrazellulärer Matrix sowie Wundkontraktion
Reifung (maturation) mit Neubildung extrazellulärer Matrix sowie Wundkontraktion  systemische:
Ernährung (z.B. Eiweiß- oder Vitaminmangel!), Stoffwechselzustand (z.B.
Diabetes mellitus!), Kreislauf (z.B. Arteriosklerose!), Hormonstatus
(z.B. Glukokortikoide!)
systemische:
Ernährung (z.B. Eiweiß- oder Vitaminmangel!), Stoffwechselzustand (z.B.
Diabetes mellitus!), Kreislauf (z.B. Arteriosklerose!), Hormonstatus
(z.B. Glukokortikoide!) lokale: Mechanische (Druck / Überdehnung an Wunde), Lage und Größe der Wunde, Fremdkörper, Infektionen
lokale: Mechanische (Druck / Überdehnung an Wunde), Lage und Größe der Wunde, Fremdkörper, Infektionen
 Western blots identifizieren Proteine: Diese Methode ermöglicht die Auftrennung von Zielproteinen nach ihrem Molekulargewicht. Antikörper
(monoklonal oder polyklonal) dienen als Sonden zur spezifischen
Bindung der Zielproteine.
Western blots identifizieren Proteine: Diese Methode ermöglicht die Auftrennung von Zielproteinen nach ihrem Molekulargewicht. Antikörper
(monoklonal oder polyklonal) dienen als Sonden zur spezifischen
Bindung der Zielproteine.
 Abbildung: Darstellung von Proteinen mittels Western-Blot
Abbildung: Darstellung von Proteinen mittels Western-Blot Abbildung).
Abbildung).  Mittels (DNA-) Polymerase-Kettenreaktion (PCR: polymerase chain reaction) lassen sich DNA-Abschnitte in vitro vervielfältigen und dadurch molekulare Sonden herstellen. Mit deren Hilfe lassen sich DNA-Sequenzen präzise orten (DNA sequencing).
Mittels (DNA-) Polymerase-Kettenreaktion (PCR: polymerase chain reaction) lassen sich DNA-Abschnitte in vitro vervielfältigen und dadurch molekulare Sonden herstellen. Mit deren Hilfe lassen sich DNA-Sequenzen präzise orten (DNA sequencing). Die PCR ermöglicht eine rasche Kopie und Multiplikation spezifischer DNA-Sequenzen bis zu einer Länge von 1 kb (103 Basen) zu analytischen und präparativen Zwecken. Die Methode nutzt kurze, Endstücken komplementäre Oligonukleotid-Primer.
Die PCR ermöglicht eine rasche Kopie und Multiplikation spezifischer DNA-Sequenzen bis zu einer Länge von 1 kb (103 Basen) zu analytischen und präparativen Zwecken. Die Methode nutzt kurze, Endstücken komplementäre Oligonukleotid-Primer. Southern blots identifizieren Gene: Es werden Nukleinsäuresequenzen (Gensonden, hybridization probes)
hergestellt (PCR), die komplementär zu der gesuchten DNA-Sequenz sind.
Mit ihrer Hilfe können Gensequenzen z.B. in Zellhomogenaten aufgefunden
werden (Einzelheiten wie Gelelektrophorese, Blotting von Gel auf
Nitrozellulose, Hybridisierung und Sichtbarmachung s. Biochemie).
Southern blots identifizieren Gene: Es werden Nukleinsäuresequenzen (Gensonden, hybridization probes)
hergestellt (PCR), die komplementär zu der gesuchten DNA-Sequenz sind.
Mit ihrer Hilfe können Gensequenzen z.B. in Zellhomogenaten aufgefunden
werden (Einzelheiten wie Gelelektrophorese, Blotting von Gel auf
Nitrozellulose, Hybridisierung und Sichtbarmachung s. Biochemie). Northern blots identifizieren mRNA-Transkripte:
Diese Methode erlaubt es festzustellen, welche Gewebe ein bestimmtes
RNA-Transkript herstellen. Das Verfahren ist ähnlich wie beim
Southern-Blot (Homogenisierung fraglicher Zellen etc), nur werden RNA-
und nicht DNA-Abschnitte nachgewiesen.
Northern blots identifizieren mRNA-Transkripte:
Diese Methode erlaubt es festzustellen, welche Gewebe ein bestimmtes
RNA-Transkript herstellen. Das Verfahren ist ähnlich wie beim
Southern-Blot (Homogenisierung fraglicher Zellen etc), nur werden RNA-
und nicht DNA-Abschnitte nachgewiesen. Immunozytochemie: Wie beim Nachweis von RNA, kann auch der Proteinnachweis - statt in Zellhomogenaten - in Gewebeschnitten
erfolgen und so die Lokalisierung der gesuchten Proteine im
untersuchten Gewebe (auch innerhalb von Zellen) sichtbar gemacht werden
- nur eben nicht mittels Hybridisierung, sondern mit Hilfe von
Antigen-Antikörper-Reaktionen.
Immunozytochemie: Wie beim Nachweis von RNA, kann auch der Proteinnachweis - statt in Zellhomogenaten - in Gewebeschnitten
erfolgen und so die Lokalisierung der gesuchten Proteine im
untersuchten Gewebe (auch innerhalb von Zellen) sichtbar gemacht werden
- nur eben nicht mittels Hybridisierung, sondern mit Hilfe von
Antigen-Antikörper-Reaktionen.

 Chromosomen bestehen aus einem vielfach verwickelten, aber unverzweigten Chromatinfaden - einem DNA-Molekül mit Begleitproteinen (Histone, Nicht-Histone). Histone regulieren die Verpackung und Ablesung der DNA. Multiples Aufspulen der Chromatinfäden ermöglicht platzsparende Verpackung (z.B. Metaphase der Mitose), in der Chromosomen kondensiert sind, allerdings kaum abgelesen werden können
Chromosomen bestehen aus einem vielfach verwickelten, aber unverzweigten Chromatinfaden - einem DNA-Molekül mit Begleitproteinen (Histone, Nicht-Histone). Histone regulieren die Verpackung und Ablesung der DNA. Multiples Aufspulen der Chromatinfäden ermöglicht platzsparende Verpackung (z.B. Metaphase der Mitose), in der Chromosomen kondensiert sind, allerdings kaum abgelesen werden können Demethylierung
der DNA und Acetylierung von Histonen bewirkt eine offene Konformation
mit ablesbaren Genen, Methylierung der DNA und Deacetylierung von
Histonen geschlossene Konformation mit
"abgeschalteten" Genen. Das Epigenom
bezieht sich auf Veränderungen am Chromatin, die nicht die Basensequenz
der DNA, sondern die Realisierung (Expression) genetischer Information
betreffen
Demethylierung
der DNA und Acetylierung von Histonen bewirkt eine offene Konformation
mit ablesbaren Genen, Methylierung der DNA und Deacetylierung von
Histonen geschlossene Konformation mit
"abgeschalteten" Genen. Das Epigenom
bezieht sich auf Veränderungen am Chromatin, die nicht die Basensequenz
der DNA, sondern die Realisierung (Expression) genetischer Information
betreffen Von einer typischen Zelle werden jeweils mehrere tausend Gene - RNA-codierende DNA-Abschnitte - ausgelesen
(transkribiert). Aktivierung von Promotorsequenzen gibt die Transkription - das Kopieren als RNA -
frei, damit beginnt die Expression (Realisierung eines Genproduktes). Die Ablesung (Transkription) von Genen erfolgt durch RNA-Polymerasen, die sich dazu an eine vor dem Gen gelegene Promotorsequenz anlagern müssen. Dazu brauchen sie DNA-bindende Proteine (Transkriptionsfaktoren). Gene können verschieden aktiviert werden, abhängig vom Muster der anregenden Faktoren. Am Transkriptionsstartpunkt müssen sich zahlreiche Proteine zusammenfinden, um die Transkription zu starten. Die Bindung regulatorischer Faktoren verändert
die Chromatinstruktur und fördert oder hemmt die Aktivität von RNA-Polymerase sowie die Bindung von Transkriptionsfaktoren
Von einer typischen Zelle werden jeweils mehrere tausend Gene - RNA-codierende DNA-Abschnitte - ausgelesen
(transkribiert). Aktivierung von Promotorsequenzen gibt die Transkription - das Kopieren als RNA -
frei, damit beginnt die Expression (Realisierung eines Genproduktes). Die Ablesung (Transkription) von Genen erfolgt durch RNA-Polymerasen, die sich dazu an eine vor dem Gen gelegene Promotorsequenz anlagern müssen. Dazu brauchen sie DNA-bindende Proteine (Transkriptionsfaktoren). Gene können verschieden aktiviert werden, abhängig vom Muster der anregenden Faktoren. Am Transkriptionsstartpunkt müssen sich zahlreiche Proteine zusammenfinden, um die Transkription zu starten. Die Bindung regulatorischer Faktoren verändert
die Chromatinstruktur und fördert oder hemmt die Aktivität von RNA-Polymerase sowie die Bindung von Transkriptionsfaktoren Es gibt Boten- (m-), ribosomale (r-), Transfer- (t-) und weitere kleine Ribonukleinsäuren (s-RNA). Nur ~1,5% des gesamten Genoms sind proteincodierende Sequenzen; im Zellkern zunächst gebildete heteronukleäre RNA (hn-RNA) wird posttranslational bearbeitet: Entfernen von (nicht-proteincodierenden) Introns und Spleißen von (proteincodierenden) Exons; m-RNA ethält die Bauanleitung für Proteine. t-RNA
ermöglichen die
Translation (Übersetzung in die Aminosäuresprache): Sie binden einerseits ein komplementäres Basentriplet der m-RNA,
andererseits bringen sie die entsprechende Aminosäure. Proteine können posttranslational modifiziert werden
Es gibt Boten- (m-), ribosomale (r-), Transfer- (t-) und weitere kleine Ribonukleinsäuren (s-RNA). Nur ~1,5% des gesamten Genoms sind proteincodierende Sequenzen; im Zellkern zunächst gebildete heteronukleäre RNA (hn-RNA) wird posttranslational bearbeitet: Entfernen von (nicht-proteincodierenden) Introns und Spleißen von (proteincodierenden) Exons; m-RNA ethält die Bauanleitung für Proteine. t-RNA
ermöglichen die
Translation (Übersetzung in die Aminosäuresprache): Sie binden einerseits ein komplementäres Basentriplet der m-RNA,
andererseits bringen sie die entsprechende Aminosäure. Proteine können posttranslational modifiziert werden Über 50
verschiedene Wachstumsfaktoren können die DNA-Synthese ihrer Zielzellen
aktivieren; diese benötigen dazu meist bestimmte
Muster an Wachstumsfaktoren. Mangel
an Wachstumsfaktorern bringt somatische Zellen in die G0-Phase oder
zur Apoptose. Die wichtigsten Wachstumsfaktoren sind BDNF
(brain-derived
neurotrophic factor), EGF (epidermal growth factor), TGF (transforming
growth factors), VEGF (vascular endothelial growth factors), NGF (nerve
growth factor), PDGF (platelet derived growth factors), FGF (fibroblast
growth factor), HGF / SF (hepatocyte growth factor / scatter factor),
IGF (insulin-like growth factors)
Über 50
verschiedene Wachstumsfaktoren können die DNA-Synthese ihrer Zielzellen
aktivieren; diese benötigen dazu meist bestimmte
Muster an Wachstumsfaktoren. Mangel
an Wachstumsfaktorern bringt somatische Zellen in die G0-Phase oder
zur Apoptose. Die wichtigsten Wachstumsfaktoren sind BDNF
(brain-derived
neurotrophic factor), EGF (epidermal growth factor), TGF (transforming
growth factors), VEGF (vascular endothelial growth factors), NGF (nerve
growth factor), PDGF (platelet derived growth factors), FGF (fibroblast
growth factor), HGF / SF (hepatocyte growth factor / scatter factor),
IGF (insulin-like growth factors)  Verletzungen
aktivieren Blutstillung, Kinin- und Immunsystem (Entzündung),
Wundverschluss, Gewebereparatur und Wundheilung. Integrine binden
extrazelluläre Komponenten und interagieren mit dem Zytoskelett an
fokalen Adhäsionskomplexen, Wachstumsfaktoren aktivieren die Transkription von Genen, die in ruhenden Zellen nicht abgelesen werden.
Es folgen Proliferation, Zellteilung, Differenzierung, Migration,
Apoptose; Angiogenese wird angeregt. Heilung erfolgt entlang
Leitstrukturen der extrazellulären Matrix (Organmorphologie), die dabei gespeicherte Wachstumsfaktoren freigibt
Verletzungen
aktivieren Blutstillung, Kinin- und Immunsystem (Entzündung),
Wundverschluss, Gewebereparatur und Wundheilung. Integrine binden
extrazelluläre Komponenten und interagieren mit dem Zytoskelett an
fokalen Adhäsionskomplexen, Wachstumsfaktoren aktivieren die Transkription von Genen, die in ruhenden Zellen nicht abgelesen werden.
Es folgen Proliferation, Zellteilung, Differenzierung, Migration,
Apoptose; Angiogenese wird angeregt. Heilung erfolgt entlang
Leitstrukturen der extrazellulären Matrix (Organmorphologie), die dabei gespeicherte Wachstumsfaktoren freigibt Die Interphase besteht aus G1-Phase (mindestens 10 Stunden: Wachstum und Aktivität der Zelle), die in eine G0-Phase münden kann, ansonsten bis zum G1-Restriktionspunkt reicht, gefolgt vom G1/S-Kontrollpunkt zur Prüfung der DNA-Integrität; S-Phase (6-8 Stunden: DNA-Reduplikation); und der
~4 Stunden dauernden G2-Phase. Am abschließenden G2/M-Checkpoint können Korrekturen an der replizierten DNA erfolgen. Die Mitose
wird durch Mitogene ausgelöst und involviert Proteinkinasen
(MAP-Kinasen), welche Mitose, Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose steuern. Die Mitose besteht aus
Pro-, Meta-, Ana- und Telophase
Die Interphase besteht aus G1-Phase (mindestens 10 Stunden: Wachstum und Aktivität der Zelle), die in eine G0-Phase münden kann, ansonsten bis zum G1-Restriktionspunkt reicht, gefolgt vom G1/S-Kontrollpunkt zur Prüfung der DNA-Integrität; S-Phase (6-8 Stunden: DNA-Reduplikation); und der
~4 Stunden dauernden G2-Phase. Am abschließenden G2/M-Checkpoint können Korrekturen an der replizierten DNA erfolgen. Die Mitose
wird durch Mitogene ausgelöst und involviert Proteinkinasen
(MAP-Kinasen), welche Mitose, Proliferation, Genexpression,
Differenzierung, Überleben und Apoptose steuern. Die Mitose besteht aus
Pro-, Meta-, Ana- und Telophase 